+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5eaj | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of DHFR in 0% Isopropanol | ||||||
 Components Components | Dihydrofolate reductase | ||||||
 Keywords Keywords | OXIDOREDUCTASE / dynamics | ||||||
| Function / homology |  Function and homology information Function and homology informationmethotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / folic acid binding / folic acid biosynthetic process / NADP+ binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity ...methotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / folic acid binding / folic acid biosynthetic process / NADP+ binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity / folic acid metabolic process / tetrahydrofolate biosynthetic process / NADPH binding / one-carbon metabolic process / NADP binding / response to xenobiotic stimulus / response to antibiotic / metal ion binding / cytosol Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.701 Å MOLECULAR REPLACEMENT / Resolution: 1.701 Å | ||||||
 Authors Authors | Cuneo, M.J. / Agarwal, P.K. | ||||||
 Citation Citation |  Journal: Biochemistry / Year: 2018 Journal: Biochemistry / Year: 2018Title: Modulating Enzyme Activity by Altering Protein Dynamics with Solvent. Authors: Duff Jr., M.R. / Borreguero, J.M. / Cuneo, M.J. / Ramanathan, A. / He, J. / Kamath, G. / Chennubhotla, S.C. / Meilleur, F. / Howell, E.E. / Herwig, K.W. / Myles, D.A.A. / Agarwal, P.K. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5eaj.cif.gz 5eaj.cif.gz | 90.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5eaj.ent.gz pdb5eaj.ent.gz | 66.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5eaj.json.gz 5eaj.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ea/5eaj https://data.pdbj.org/pub/pdb/validation_reports/ea/5eaj ftp://data.pdbj.org/pub/pdb/validation_reports/ea/5eaj ftp://data.pdbj.org/pub/pdb/validation_reports/ea/5eaj | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5ujxC  1rd7S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 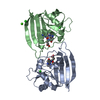
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 18020.326 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: folA, AA953_06220, ABE89_23390, ABE90_01590, ABE91_11465, ABE92_27580, ABE93_03540, ABE94_26505, ABE95_19105, AC789_1c00510, ACM16_01115, ACM17_00910, ACM18_00225, ACM19_01115, ACM20_00925, ...Gene: folA, AA953_06220, ABE89_23390, ABE90_01590, ABE91_11465, ABE92_27580, ABE93_03540, ABE94_26505, ABE95_19105, AC789_1c00510, ACM16_01115, ACM17_00910, ACM18_00225, ACM19_01115, ACM20_00925, ACM21_22820, BN1008_2275, ECONIH1_00280, ECs0051, EL75_3711, EL79_3822, EL80_3768, HW43_03985, IY32_08385, JD73_13725, NMECO18_25965, PD07_16380, PU06_14795, PU15_01135, PU21_08285, PU33_00085, PU38_11910, PU53_21230, PU69_06270, PU71_09850, PU73_17465, RR31_00265, SK64_04362, SK65_04102, SK81_04106, SY51_00265, TZ57_00270, UC21_17710, UC41_15235, UN86_27670, UN92_06125, WQ64_08430, WQ65_17175, WQ66_04025, WQ69_03315, WQ70_09735, WQ71_08985, WQ72_04470, WQ73_14425, WQ74_13220, WQ77_00770, WQ79_09130, WQ84_04435, WQ86_16125, WQ88_00770, WQ91_15025, WQ92_19155, WQ95_08145, WQ99_15285, WR00_12370, WR01_07485, WR03_05655, WR04_00040, WR05_03890, WR12_01840, WR13_10745, WR16_15930, WR17_04125, WR18_06540, WR19_18465, WR23_10335, WR24_10640, XB00_01295, XB01_19915 Production host:  References: UniProt: C3TR70, UniProt: P0ABQ4*PLUS, dihydrofolate reductase #2: Chemical | #3: Chemical | ChemComp-CA / | #4: Chemical | ChemComp-CL / #5: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.47 Å3/Da / Density % sol: 50.3 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 7.5 Details: 0.2 M CaCl2, 20-35 % (w/v) PEG 6000, 0.1M NaHEPES pH7.5 |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 19-ID / Wavelength: 1 Å / Beamline: 19-ID / Wavelength: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: MAR scanner 345 mm plate / Detector: IMAGE PLATE / Date: Feb 1, 2012 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.7→40 Å / Num. all: 38279 / Num. obs: 38279 / % possible obs: 99.1 % / Redundancy: 8.4 % / Biso Wilson estimate: 22.79 Å2 / Rmerge(I) obs: 0.061 / Χ2: 1.357 / Net I/av σ(I): 38.455 / Net I/σ(I): 15.8 / Num. measured all: 320420 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 1RD7 Resolution: 1.701→38.916 Å / FOM work R set: 0.8315 / SU ML: 0.23 / Cross valid method: FREE R-VALUE / σ(F): 1.38 / Phase error: 24.32 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL / Bsol: 35.42 Å2 / ksol: 0.359 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 104.52 Å2 / Biso mean: 30.72 Å2 / Biso min: 9.81 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.701→38.916 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 14
|
 Movie
Movie Controller
Controller













 PDBj
PDBj