[English] 日本語
 Yorodumi
Yorodumi- PDB-5dal: Crystal Structure of human Glutathione Transferase Pi complexed w... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5dal | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of human Glutathione Transferase Pi complexed with a metalloid in the presence of Glutathione | ||||||
 Components Components | (Glutathione S-transferase ...) x 2 | ||||||
 Keywords Keywords | TRANSFERASE / Metalloid / Anti-cancer | ||||||
| Function / homology |  Function and homology information Function and homology informationS-nitrosoglutathione binding / nitric oxide storage / negative regulation of leukocyte proliferation / TRAF2-GSTP1 complex / negative regulation of smooth muscle cell chemotaxis / dinitrosyl-iron complex binding / common myeloid progenitor cell proliferation / hepoxilin biosynthetic process / cellular response to cell-matrix adhesion / glutathione derivative biosynthetic process ...S-nitrosoglutathione binding / nitric oxide storage / negative regulation of leukocyte proliferation / TRAF2-GSTP1 complex / negative regulation of smooth muscle cell chemotaxis / dinitrosyl-iron complex binding / common myeloid progenitor cell proliferation / hepoxilin biosynthetic process / cellular response to cell-matrix adhesion / glutathione derivative biosynthetic process / response to L-ascorbic acid / linoleic acid metabolic process / Glutathione conjugation / negative regulation of monocyte chemotactic protein-1 production / nitric oxide binding / JUN kinase binding / glutathione peroxidase activity / Paracetamol ADME / oligodendrocyte development / negative regulation of stress-activated MAPK cascade / negative regulation of JNK cascade / prostaglandin metabolic process / cellular response to glucocorticoid stimulus / negative regulation of interleukin-1 beta production / regulation of stress-activated MAPK cascade / Detoxification of Reactive Oxygen Species / negative regulation of acute inflammatory response / glutathione transferase / negative regulation of vascular associated smooth muscle cell proliferation / glutathione transferase activity / negative regulation of tumor necrosis factor production / animal organ regeneration / protein serine/threonine kinase inhibitor activity / response to amino acid / negative regulation of tumor necrosis factor-mediated signaling pathway / toxic substance binding / regulation of ERK1 and ERK2 cascade / negative regulation of fibroblast proliferation / negative regulation of MAPK cascade / positive regulation of superoxide anion generation / glutathione metabolic process / xenobiotic metabolic process / cellular response to epidermal growth factor stimulus / fatty acid binding / negative regulation of canonical NF-kappaB signal transduction / central nervous system development / response to reactive oxygen species / negative regulation of extrinsic apoptotic signaling pathway / negative regulation of ERK1 and ERK2 cascade / cellular response to insulin stimulus / response to estradiol / cellular response to lipopolysaccharide / secretory granule lumen / vesicle / ficolin-1-rich granule lumen / response to ethanol / Neutrophil degranulation / negative regulation of apoptotic process / negative regulation of transcription by RNA polymerase II / mitochondrion / extracellular space / extracellular exosome / extracellular region / nucleus / cytoplasm / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.5 Å molecular replacement / Resolution: 1.5 Å | ||||||
 Authors Authors | Parker, L.J. / Parker, M.W. / Morton, C.J. | ||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Visualisation of Organoarsenic Human Glutathione Transferase P1-1 Complexes: Metabolism of Arsenic-based Therapeutics Authors: Parker, L.J. / Parker, M.W. / Morton, C.J. / Bocedi, A. / Ascher, D.B. / Aitken, J.B. / Harris, H.H. / Lo Bello, M. / Ricci, G. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5dal.cif.gz 5dal.cif.gz | 187.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5dal.ent.gz pdb5dal.ent.gz | 149.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5dal.json.gz 5dal.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/da/5dal https://data.pdbj.org/pub/pdb/validation_reports/da/5dal ftp://data.pdbj.org/pub/pdb/validation_reports/da/5dal ftp://data.pdbj.org/pub/pdb/validation_reports/da/5dal | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5dakC  5dcgC  5ddlC  5gssS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
|
- Components
Components
-Glutathione S-transferase ... , 2 types, 2 molecules AB
| #1: Protein | Mass: 23393.770 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: GSTP1, FAEES3, GST3 / Production host: Homo sapiens (human) / Gene: GSTP1, FAEES3, GST3 / Production host:  |
|---|---|
| #2: Protein | Mass: 23377.770 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: GSTP1, FAEES3, GST3 / Production host: Homo sapiens (human) / Gene: GSTP1, FAEES3, GST3 / Production host:  |
-Non-polymers , 6 types, 489 molecules 
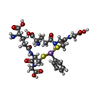

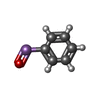




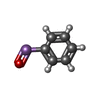


| #3: Chemical | | #4: Chemical | #5: Chemical | #6: Chemical | #7: Chemical | ChemComp-SO4 / | #8: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.54 Å3/Da / Density % sol: 51.63 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 6 Details: 100MM MES, PH 6.0, 28% (W/V) PEG 8000, 20MM CACL2, 10MM DTT, 10MM GSH. Soaked in 2MM PAO dissolved in DMSO |
-Data collection
| Diffraction | Mean temperature: 100 K | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 14-BM-C / Wavelength: 0.9 Å / Beamline: 14-BM-C / Wavelength: 0.9 Å | |||||||||||||||||||||||||||
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Nov 17, 2005 | |||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9 Å / Relative weight: 1 | |||||||||||||||||||||||||||
| Reflection | Resolution: 1.5→35.63 Å / Num. obs: 74367 / % possible obs: 99.5 % / Redundancy: 6.8 % / Biso Wilson estimate: 14.99 Å2 / CC1/2: 0.995 / Rmerge(I) obs: 0.076 / Rpim(I) all: 0.032 / Net I/σ(I): 15.2 / Num. measured all: 508961 / Scaling rejects: 5242 | |||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
-Phasing
| Phasing | Method:  molecular replacement molecular replacement | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR |
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB entry 5GSS Resolution: 1.5→34 Å / FOM work R set: 0.8681 / SU ML: 0.16 / Cross valid method: FREE R-VALUE / σ(F): 1.38 / Phase error: 21.07 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 80.15 Å2 / Biso mean: 21.36 Å2 / Biso min: 6.77 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.5→34 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 26
|
 Movie
Movie Controller
Controller












 PDBj
PDBj









