| 登録情報 | データベース: PDB / ID: 5c7u
|
|---|
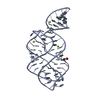
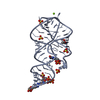
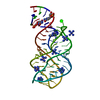
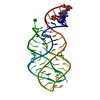
| タイトル | 5'-monophosphate wt Guanine Riboswitch bound to hypoxanthine. |
|---|
 要素 要素 | 5'-monophosphate wt guanine riboswitch |
|---|
 キーワード キーワード | RNA / expanded RNA / riboswitch / Unnatural base pair |
|---|
| 機能・相同性 | HYPOXANTHINE / COBALT HEXAMMINE(III) / : / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Bacillus subtilis subsp. subtilis str. 168 (枯草菌) Bacillus subtilis subsp. subtilis str. 168 (枯草菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.05 Å 分子置換 / 解像度: 3.05 Å |
|---|
 データ登録者 データ登録者 | Hernandez, A.R. / Shao, Y. / Hoshika, S. / Yang, Z. / Shelke, S.A. / Herrou, J. / Kim, H.-J. / Kim, M.-J. / Piccirilli, J.A. / Benner, S.A. |
|---|
| 資金援助 |  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Defense Threat Reduction Agency (DTRA) | HDTRA1-13-1-0004 |  米国 米国 | | National Aeronautic Space Administration (NASA, United States) | NNX10AT28G |  米国 米国 | | Templeton World Charitable Fund | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2015 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2015
タイトル: A Crystal Structure of a Functional RNA Molecule Containing an Artificial Nucleobase Pair.
著者: Hernandez, A.R. / Shao, Y. / Hoshika, S. / Yang, Z. / Shelke, S.A. / Herrou, J. / Kim, H.J. / Kim, M.J. / Piccirilli, J.A. / Benner, S.A. |
|---|
| 履歴 | | 登録 | 2015年6月24日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年8月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年8月19日 | Group: Database references |
|---|
| 改定 1.2 | 2020年1月1日 | Group: Author supporting evidence / Database references / Derived calculations
カテゴリ: citation / pdbx_audit_support / pdbx_struct_oper_list
Item: _citation.journal_id_CSD / _pdbx_audit_support.funding_organization / _pdbx_struct_oper_list.symmetry_operation |
|---|
| 改定 1.3 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.05 Å
分子置換 / 解像度: 3.05 Å  データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2015
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5c7u.cif.gz
5c7u.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5c7u.ent.gz
pdb5c7u.ent.gz PDB形式
PDB形式 5c7u.json.gz
5c7u.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5c7u_validation.pdf.gz
5c7u_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5c7u_full_validation.pdf.gz
5c7u_full_validation.pdf.gz 5c7u_validation.xml.gz
5c7u_validation.xml.gz 5c7u_validation.cif.gz
5c7u_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/c7/5c7u
https://data.pdbj.org/pub/pdb/validation_reports/c7/5c7u ftp://data.pdbj.org/pub/pdb/validation_reports/c7/5c7u
ftp://data.pdbj.org/pub/pdb/validation_reports/c7/5c7u リンク
リンク 集合体
集合体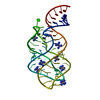
 要素
要素
 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 24-ID-C / 波長: 0.979 Å
/ ビームライン: 24-ID-C / 波長: 0.979 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj