[English] 日本語
 Yorodumi
Yorodumi- PDB-5bqe: Crystal structure of Norrin in complex with the cysteine-rich dom... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5bqe | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of Norrin in complex with the cysteine-rich domain of Frizzled 4 -Methylated form | ||||||||||||
 Components Components |
| ||||||||||||
 Keywords Keywords | SIGNALING PROTEIN / Wnt signalling pathway / Norrie disease protein / Glycoprotein / G protein coupled receptor | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationcerebellum vasculature morphogenesis / Wnt signaling pathway, calcium modulating pathway / retina blood vessel maintenance / cone retinal bipolar cell differentiation / Norrin signaling pathway / extracellular matrix-cell signaling / progesterone secretion / retinal rod cell differentiation / re-entry into mitotic cell cycle / retinal blood vessel morphogenesis ...cerebellum vasculature morphogenesis / Wnt signaling pathway, calcium modulating pathway / retina blood vessel maintenance / cone retinal bipolar cell differentiation / Norrin signaling pathway / extracellular matrix-cell signaling / progesterone secretion / retinal rod cell differentiation / re-entry into mitotic cell cycle / retinal blood vessel morphogenesis / retina vasculature morphogenesis in camera-type eye / locomotion involved in locomotory behavior / Signaling by RNF43 mutants / WNT5A-dependent internalization of FZD4 / glycine metabolic process / regulation of vascular endothelial growth factor receptor signaling pathway / retina layer formation / positive regulation of neuron projection arborization / Wnt receptor activity / retinal pigment epithelium development / non-canonical Wnt signaling pathway / microglial cell proliferation / endothelial cell differentiation / Wnt-protein binding / dendritic spine development / establishment of blood-retinal barrier / vacuole organization / protein targeting to lysosome / microglia differentiation / establishment of blood-brain barrier / frizzled binding / optic nerve development / positive regulation of dendrite morphogenesis / Class B/2 (Secretin family receptors) / cytokine receptor activity / retinal ganglion cell axon guidance / lens development in camera-type eye / ubiquitin-dependent endocytosis / smoothened signaling pathway / cytokine binding / exploration behavior / decidualization / action potential / negative regulation of cell-substrate adhesion / blood vessel remodeling / canonical Wnt signaling pathway / response to axon injury / vasculogenesis / tricarboxylic acid cycle / cellular response to retinoic acid / glutathione metabolic process / visual perception / substrate adhesion-dependent cell spreading / transforming growth factor beta receptor signaling pathway / Regulation of FZD by ubiquitination / cytokine activity / cellular response to leukemia inhibitory factor / PDZ domain binding / Asymmetric localization of PCP proteins / clathrin-coated endocytic vesicle membrane / sensory perception of sound / G protein-coupled receptor activity / : / Wnt signaling pathway / neuron differentiation / cell-cell junction / nervous system development / Cargo recognition for clathrin-mediated endocytosis / mitotic cell cycle / signaling receptor activity / amyloid-beta binding / Clathrin-mediated endocytosis / neuron apoptotic process / Ca2+ pathway / angiogenesis / cellular response to hypoxia / transcription by RNA polymerase II / response to hypoxia / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / cell population proliferation / cilium / positive regulation of cell migration / protein ubiquitination / inflammatory response / protein heterodimerization activity / ubiquitin protein ligase binding / dendrite / positive regulation of DNA-templated transcription / protein-containing complex binding / glutamatergic synapse / cell surface / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / extracellular space / nucleoplasm / plasma membrane Similarity search - Function | ||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 2.3 Å molecular replacement / Resolution: 2.3 Å | ||||||||||||
 Authors Authors | Chang, T.-H. / Hsieh, F.-L. / Harlos, K. / Jones, E.Y. | ||||||||||||
| Funding support |  United Kingdom, 3items United Kingdom, 3items
| ||||||||||||
 Citation Citation |  Journal: Elife / Year: 2015 Journal: Elife / Year: 2015Title: Structure and functional properties of Norrin mimic Wnt for signalling with Frizzled4, Lrp5/6, and proteoglycan. Authors: Chang, T.H. / Hsieh, F.L. / Zebisch, M. / Harlos, K. / Elegheert, J. / Jones, E.Y. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5bqe.cif.gz 5bqe.cif.gz | 88.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5bqe.ent.gz pdb5bqe.ent.gz | 65.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5bqe.json.gz 5bqe.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqe https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqe ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqe ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqe | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5bpbSC 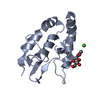 5bpqC  5bpuSC  5bq8C  5bqbC  5bqcC C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 3 types, 3 molecules ABC
| #1: Protein | Mass: 13614.706 Da / Num. of mol.: 1 / Fragment: residues 25-133 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: NDP, EVR2 / Plasmid: pHLIgK-STR-8H-SUMO-1D4 / Cell line (production host): HEK293T / Production host: Homo sapiens (human) / Gene: NDP, EVR2 / Plasmid: pHLIgK-STR-8H-SUMO-1D4 / Cell line (production host): HEK293T / Production host:  Homo sapiens (human) / References: UniProt: Q00604 Homo sapiens (human) / References: UniProt: Q00604 |
|---|---|
| #2: Protein | Mass: 13722.891 Da / Num. of mol.: 1 / Fragment: residues 25-133 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: NDP, EVR2 / Plasmid: pHLIgK-STR-8H-SUMO-1D4 / Cell line (production host): HEK293T / Production host: Homo sapiens (human) / Gene: NDP, EVR2 / Plasmid: pHLIgK-STR-8H-SUMO-1D4 / Cell line (production host): HEK293T / Production host:  Homo sapiens (human) / References: UniProt: Q00604 Homo sapiens (human) / References: UniProt: Q00604 |
| #3: Protein | Mass: 16759.324 Da / Num. of mol.: 1 / Fragment: residues 42-179 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: FZD4 / Plasmid: pHLsec-mVenus-12H / Cell line (production host): HEK293T / Production host: Homo sapiens (human) / Gene: FZD4 / Plasmid: pHLsec-mVenus-12H / Cell line (production host): HEK293T / Production host:  Homo sapiens (human) / References: UniProt: Q9ULV1 Homo sapiens (human) / References: UniProt: Q9ULV1 |
-Sugars , 1 types, 1 molecules 
| #5: Sugar | ChemComp-NAG / |
|---|
-Non-polymers , 3 types, 119 molecules 




| #4: Chemical | | #6: Chemical | ChemComp-CL / | #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.34 Å3/Da / Density % sol: 63.18 % |
|---|---|
| Crystal grow | Temperature: 294 K / Method: vapor diffusion, sitting drop / Details: 0.1 M Bicine, pH 9.0, 10% PEG6000 |
-Data collection
| Diffraction | Mean temperature: 100 K | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I04 / Wavelength: 0.97949 Å / Beamline: I04 / Wavelength: 0.97949 Å | |||||||||||||||||||||||||||
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Sep 15, 2013 | |||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.97949 Å / Relative weight: 1 | |||||||||||||||||||||||||||
| Reflection | Resolution: 2.3→49.46 Å / Num. obs: 26816 / % possible obs: 98.9 % / Redundancy: 6 % / Biso Wilson estimate: 48.29 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.103 / Rpim(I) all: 0.045 / Net I/σ(I): 10.7 / Num. measured all: 161410 / Scaling rejects: 9 | |||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
-Phasing
| Phasing | Method:  molecular replacement molecular replacement | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR | Model details: Phaser MODE: MR_AUTO
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5BPB,5BPU Resolution: 2.3→49.46 Å / FOM work R set: 0.7608 / SU ML: 0.32 / Cross valid method: THROUGHOUT / σ(F): 1.34 / Phase error: 23.04 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 174.26 Å2 / Biso mean: 63.08 Å2 / Biso min: 30.05 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.3→49.46 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 10
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj








