| 登録情報 | データベース: PDB / ID: 5b4o
|
|---|
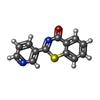
| タイトル | Crystal structure of Macrophage Migration Inhibitory Factor in complex with BTZO-14 |
|---|
 要素 要素 | Macrophage migration inhibitory factor |
|---|
 キーワード キーワード | ISOMERASE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of prostaglandin secretion involved in immune response / positive regulation of myeloid leukocyte cytokine production involved in immune response / phenylpyruvate tautomerase / L-dopachrome isomerase / dopachrome isomerase activity / phenylpyruvate tautomerase activity / positive regulation of lipopolysaccharide-mediated signaling pathway / cytokine receptor binding / positive regulation of arachidonic acid secretion / negative regulation of myeloid cell apoptotic process ...positive regulation of prostaglandin secretion involved in immune response / positive regulation of myeloid leukocyte cytokine production involved in immune response / phenylpyruvate tautomerase / L-dopachrome isomerase / dopachrome isomerase activity / phenylpyruvate tautomerase activity / positive regulation of lipopolysaccharide-mediated signaling pathway / cytokine receptor binding / positive regulation of arachidonic acid secretion / negative regulation of myeloid cell apoptotic process / negative regulation of macrophage chemotaxis / negative regulation of mature B cell apoptotic process / carboxylic acid metabolic process / positive regulation of chemokine (C-X-C motif) ligand 2 production / prostaglandin biosynthetic process / negative regulation of protein metabolic process / regulation of macrophage activation / negative regulation of intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / chemoattractant activity / positive regulation of protein kinase A signaling / protein homotrimerization / negative regulation of cellular senescence / negative regulation of DNA damage response, signal transduction by p53 class mediator / DNA damage response, signal transduction by p53 class mediator / positive regulation of phosphorylation / positive regulation of B cell proliferation / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / negative regulation of cell migration / positive regulation of cytokine production / cytokine activity / Cell surface interactions at the vascular wall / positive regulation of tumor necrosis factor production / positive regulation of peptidyl-tyrosine phosphorylation / cellular senescence / positive regulation of fibroblast proliferation / positive regulation of peptidyl-serine phosphorylation / secretory granule lumen / protease binding / vesicle / ficolin-1-rich granule lumen / positive regulation of ERK1 and ERK2 cascade / cell surface receptor signaling pathway / inflammatory response / negative regulation of gene expression / innate immune response / positive regulation of cell population proliferation / Neutrophil degranulation / negative regulation of apoptotic process / cell surface / extracellular space / extracellular exosome / extracellular region / nucleoplasm / identical protein binding / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 Macrophage migration inhibitory factor, conserved site / Macrophage migration inhibitory factor family signature. / Macrophage migration inhibitory factor / Macrophage migration inhibitory factor (MIF) / Macrophage Migration Inhibitory Factor / Macrophage Migration Inhibitory Factor / Tautomerase/MIF superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 2-pyridin-3-yl-1,3-benzothiazin-4-one / Macrophage migration inhibitory factor類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.37 Å 分子置換 / 解像度: 1.37 Å |
|---|
 データ登録者 データ登録者 | Oki, H. / Igaki, S. / Moriya, Y. / Hayano, Y. / Habuka, N. |
|---|
 引用 引用 |  ジャーナル: Chem. Biol. / 年: 2010 ジャーナル: Chem. Biol. / 年: 2010
タイトル: BTZO-1, a cardioprotective agent, reveals that macrophage migration inhibitory factor regulates ARE-mediated gene expression
著者: Kimura, H. / Sato, Y. / Tajima, Y. / Suzuki, H. / Yukitake, H. / Imaeda, T. / Kajino, M. / Oki, H. / Takizawa, M. / Tanida, S. |
|---|
| 履歴 | | 登録 | 2016年4月7日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2016年4月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年11月8日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_oper_list.symmetry_operation |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.37 Å
分子置換 / 解像度: 1.37 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chem. Biol. / 年: 2010
ジャーナル: Chem. Biol. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5b4o.cif.gz
5b4o.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5b4o.ent.gz
pdb5b4o.ent.gz PDB形式
PDB形式 5b4o.json.gz
5b4o.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5b4o_validation.pdf.gz
5b4o_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5b4o_full_validation.pdf.gz
5b4o_full_validation.pdf.gz 5b4o_validation.xml.gz
5b4o_validation.xml.gz 5b4o_validation.cif.gz
5b4o_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/b4/5b4o
https://data.pdbj.org/pub/pdb/validation_reports/b4/5b4o ftp://data.pdbj.org/pub/pdb/validation_reports/b4/5b4o
ftp://data.pdbj.org/pub/pdb/validation_reports/b4/5b4o
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: MIF, GLIF, MMIF / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MIF, GLIF, MMIF / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.3 / 波長: 1 Å
/ ビームライン: 5.0.3 / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj