| 登録情報 | データベース: PDB / ID: 4z02
|
|---|
| タイトル | Crystal structure of BRD1 in complex with Isoquinoline-3-carboxylic acid |
|---|
 要素 要素 | Bromodomain-containing protein 1 |
|---|
 キーワード キーワード | HISTONE-BINDING PROTEIN / BRPF2 Fragment / BRD1 / Structural Genomics / Structural Genomics Consortium / SGC |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
unmodified histone reader activity / histone H3-K14 acetyltransferase complex / MOZ/MORF histone acetyltransferase complex / regulation of developmental process / regulation of hemopoiesis / erythrocyte maturation / histone reader activity / response to immobilization stress / response to electrical stimulus / Regulation of TP53 Activity through Acetylation ...unmodified histone reader activity / histone H3-K14 acetyltransferase complex / MOZ/MORF histone acetyltransferase complex / regulation of developmental process / regulation of hemopoiesis / erythrocyte maturation / histone reader activity / response to immobilization stress / response to electrical stimulus / Regulation of TP53 Activity through Acetylation / positive regulation of erythrocyte differentiation / HATs acetylate histones / perikaryon / nuclear speck / chromatin remodeling / dendrite / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / zinc ion binding / nucleus類似検索 - 分子機能 BRPF2, ePHD domain / BRPF2, PHD domain / SH3 type barrels. - #140 / : / Enhancer of polycomb-like, N-terminal / Enhancer of polycomb-like / PHD-finger / PHD-zinc-finger like domain / Extended PHD (ePHD) domain / Extended PHD (ePHD) domain profile. ...BRPF2, ePHD domain / BRPF2, PHD domain / SH3 type barrels. - #140 / : / Enhancer of polycomb-like, N-terminal / Enhancer of polycomb-like / PHD-finger / PHD-zinc-finger like domain / Extended PHD (ePHD) domain / Extended PHD (ePHD) domain profile. / domain with conserved PWWP motif / PWWP domain / PWWP domain profile. / PWWP domain / Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-finger / Zinc finger, PHD-type / PHD zinc finger / Zinc finger, FYVE/PHD-type / SH3 type barrels. / Bromodomain, conserved site / Bromodomain signature. / Bromodomain / bromo domain / Bromodomain / Bromodomain (BrD) profile. / Bromodomain-like superfamily / Zinc finger, RING/FYVE/PHD-type / Roll / Mainly Beta類似検索 - ドメイン・相同性 isoquinoline-3-carboxylic acid / Bromodomain-containing protein 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.87 Å 分子置換 / 解像度: 1.87 Å |
|---|
 データ登録者 データ登録者 | DONG, A. / IQBAL, A. / WALKER, J.R. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / BROWN, P.J. / Structural Genomics Consortium (SGC) |
|---|
 引用 引用 |  ジャーナル: to be published ジャーナル: to be published
タイトル: Crystal structure of BRD1 incomplex with Isoquinoline-3-carboxylic acid
著者: IQBAL, A. / DONG, A. / WALKER, J.R. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / BROWN, P.J. / Structural Genomics Consortium (SGC) |
|---|
| 履歴 | | 登録 | 2015年3月25日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年4月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年9月27日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Source and taxonomy
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / entity_src_gen / pdbx_initial_refinement_model / pdbx_prerelease_seq / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _entity_src_gen.pdbx_alt_source_flag / _pdbx_struct_oper_list.symmetry_operation |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.87 Å
分子置換 / 解像度: 1.87 Å  データ登録者
データ登録者 引用
引用 ジャーナル: to be published
ジャーナル: to be published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4z02.cif.gz
4z02.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4z02.ent.gz
pdb4z02.ent.gz PDB形式
PDB形式 4z02.json.gz
4z02.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4z02_validation.pdf.gz
4z02_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4z02_full_validation.pdf.gz
4z02_full_validation.pdf.gz 4z02_validation.xml.gz
4z02_validation.xml.gz 4z02_validation.cif.gz
4z02_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/z0/4z02
https://data.pdbj.org/pub/pdb/validation_reports/z0/4z02 ftp://data.pdbj.org/pub/pdb/validation_reports/z0/4z02
ftp://data.pdbj.org/pub/pdb/validation_reports/z0/4z02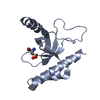
 リンク
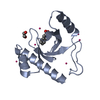
リンク 集合体
集合体

 要素
要素 Homo sapiens (ヒト) / 遺伝子: BRD1, BRL, BRPF2 / プラスミド: pET28-MHL / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: BRD1, BRL, BRPF2 / プラスミド: pET28-MHL / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-ID / 波長: 0.97918 Å
/ ビームライン: 19-ID / 波長: 0.97918 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



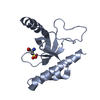









 PDBj
PDBj












