| 登録情報 | データベース: PDB / ID: 4x4v
|
|---|
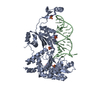
| タイトル | Crystal structure of the A.fulgidus CCA-adding enzyme in complex with a human MenBeta minihelix ending in CCACC and AMPcPP |
|---|
 要素 要素 | - CCA-adding enzyme
- Human MenBeta minihelix ending in CCACC
|
|---|
 キーワード キーワード | RNA BINDING PROTEIN / protein-RNA complex / tRNA / non-coding RNA / CCA-adding enzyme / nucleotidyltransferase / ncRNA |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
CCA tRNA nucleotidyltransferase / tRNA surveillance / CCACCA tRNA nucleotidyltransferase activity / CCA tRNA nucleotidyltransferase activity / tRNA 3'-terminal CCA addition / RNA repair / tRNA binding / magnesium ion binding / ATP binding類似検索 - 分子機能 CCA tRNA nucleotidyltransferase, domain 2 / Archaeal tRNA CCA-adding enzyme catalytic domain / tRNA nucleotidyltransferase, archaea / tRNA nucleotidyltransferase, substrate binding / tRNA nucleotidyltransferase, domain 2 / : / tRNA nucleotidyltransferase, second domain / CCA-adding enzyme, C-terminal domain / Poly(A) polymerase predicted RNA binding domain / Nucleotidyltransferase, class I-like, C-terminal ...CCA tRNA nucleotidyltransferase, domain 2 / Archaeal tRNA CCA-adding enzyme catalytic domain / tRNA nucleotidyltransferase, archaea / tRNA nucleotidyltransferase, substrate binding / tRNA nucleotidyltransferase, domain 2 / : / tRNA nucleotidyltransferase, second domain / CCA-adding enzyme, C-terminal domain / Poly(A) polymerase predicted RNA binding domain / Nucleotidyltransferase, class I-like, C-terminal / 2-5OAS/ClassI-CCAase, nucleotidyltransferase domain / Polymerase, nucleotidyl transferase domain / Poly(a)-polymerase, middle domain / Nucleotidyltransferase domain / Beta Polymerase, domain 2 / Beta Polymerase; domain 2 / Nucleotidyltransferase superfamily / Alpha-Beta Plaits / 2-Layer Sandwich / Orthogonal Bundle / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性 GUANOSINE-5'-MONOPHOSPHATE / DIPHOSPHOMETHYLPHOSPHONIC ACID ADENOSYL ESTER / D(-)-TARTARIC ACID / RNA / RNA (> 10) / CCA-adding enzyme類似検索 - 構成要素 |
|---|
| 生物種 |   Archaeoglobus fulgidus (古細菌) Archaeoglobus fulgidus (古細菌)
 Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å |
|---|
 データ登録者 データ登録者 | Kuhn, C.-D. / Joshua-Tor, L. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| Howard Hughes Medical Institute (HHMI) | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015
タイトル: On-Enzyme Refolding Permits Small RNA and tRNA Surveillance by the CCA-Adding Enzyme.
著者: Kuhn, C.D. / Wilusz, J.E. / Zheng, Y. / Beal, P.A. / Joshua-Tor, L. |
|---|
| 履歴 | | 登録 | 2014年12月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年2月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年2月18日 | Group: Database references |
|---|
| 改定 1.2 | 2015年2月25日 | Group: Database references |
|---|
| 改定 1.3 | 2017年11月22日 | Group: Derived calculations / Refinement description / Source and taxonomy
カテゴリ: entity_src_gen / pdbx_entity_src_syn ...entity_src_gen / pdbx_entity_src_syn / pdbx_struct_oper_list / software
Item: _entity_src_gen.pdbx_alt_source_flag / _pdbx_entity_src_syn.pdbx_alt_source_flag / _pdbx_struct_oper_list.symmetry_operation |
|---|
| 改定 1.4 | 2019年11月20日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.5 | 2023年12月27日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_label_asym_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Archaeoglobus fulgidus (古細菌)
Archaeoglobus fulgidus (古細菌) Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.6 Å
分子置換 / 解像度: 2.6 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Cell / 年: 2015
ジャーナル: Cell / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4x4v.cif.gz
4x4v.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4x4v.ent.gz
pdb4x4v.ent.gz PDB形式
PDB形式 4x4v.json.gz
4x4v.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4x4v_validation.pdf.gz
4x4v_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4x4v_full_validation.pdf.gz
4x4v_full_validation.pdf.gz 4x4v_validation.xml.gz
4x4v_validation.xml.gz 4x4v_validation.cif.gz
4x4v_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/x4/4x4v
https://data.pdbj.org/pub/pdb/validation_reports/x4/4x4v ftp://data.pdbj.org/pub/pdb/validation_reports/x4/4x4v
ftp://data.pdbj.org/pub/pdb/validation_reports/x4/4x4v リンク
リンク 集合体
集合体


 要素
要素
 Archaeoglobus fulgidus (古細菌) / 株: ATCC 49558 / VC-16 / DSM 4304 / JCM 9628 / NBRC 100126 / 遺伝子: cca, AF_2156 / プラスミド: pET22 / 発現宿主:
Archaeoglobus fulgidus (古細菌) / 株: ATCC 49558 / VC-16 / DSM 4304 / JCM 9628 / NBRC 100126 / 遺伝子: cca, AF_2156 / プラスミド: pET22 / 発現宿主: 
 Homo sapiens (ヒト)
Homo sapiens (ヒト)









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X25 / 波長: 1.1 Å
/ ビームライン: X25 / 波長: 1.1 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 2.6→29.68 Å / FOM work R set: 0.8376 / SU ML: 0.3 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 22.98 / 立体化学のターゲット値: ML
分子置換 / 解像度: 2.6→29.68 Å / FOM work R set: 0.8376 / SU ML: 0.3 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 22.98 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj





































