+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4ubp | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURE OF BACILLUS PASTEURII UREASE INHIBITED WITH ACETOHYDROXAMIC ACID AT 1.55 A RESOLUTION | ||||||
 要素 要素 | (PROTEIN (UREASE (CHAIN ...) x 3 | ||||||
 キーワード キーワード | HYDROLASE / UREASE / BACILLUS PASTEURII / NICKEL / ACETOHYDROXAMIC ACID / METALLOENZYME | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報urease complex / urease / urease activity / urea catabolic process / nickel cation binding / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.55 Å 分子置換 / 解像度: 1.55 Å | ||||||
 データ登録者 データ登録者 | Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Ciurli, S. / Mangani, S. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Inorg.Chem. / 年: 2000 ジャーナル: J.Biol.Inorg.Chem. / 年: 2000タイトル: The complex of Bacillus pasteurii urease with acetohydroxamate anion from X-ray data at 1.55 A resolution. 著者: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Miletti, S. / Ciurli, S. / Mangani, S. #1:  ジャーナル: Structure / 年: 1999 ジャーナル: Structure / 年: 1999タイトル: A new proposal for urease mechanism based on the crystal structures of the native and inhibited enzyme from Bacillus pasteurii: why urea hydrolysis costs two nickels. 著者: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Miletti, S. / Ciurli, S. / Mangani, S. #2: ジャーナル: Acta Crystallogr.,Sect.D / 年: 1998 タイトル: Crystallization and preliminary high-resolution X-ray diffraction analysis of native and beta-mercaptoethanol-inhibited urease from Bacillus pasteurii. 著者: Benini, S. / Ciurli, S. / Rypniewski, W.R. / Wilson, K.S. / Mangani, S. #3:  ジャーナル: J.Biol.Inorg.Chem. / 年: 1998 ジャーナル: J.Biol.Inorg.Chem. / 年: 1998タイトル: The Complex of Bacillus pasteurii Urease with Beta-Mercaptoethanol from X-Ray Data at 1.65 A Resolution 著者: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Ciurli, S. / Mangani, S. #4:  ジャーナル: Soil Biol.Biochem. / 年: 1996 ジャーナル: Soil Biol.Biochem. / 年: 1996タイトル: Bacillus pasteurii Urease: A Heteropolimeric Enzyme with a Binuclear Nickel Active Site 著者: Benini, S. / Gessa, C. / Ciurli, S. #5: ジャーナル: Eur.J.Biochem. / 年: 1996 タイトル: X-ray absorption spectroscopy study of native and phenylphosphorodiamidate-inhibited Bacillus pasteurii urease. 著者: Benini, S. / Ciurli, S. / Nolting, H.F. / Mangani, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4ubp.cif.gz 4ubp.cif.gz | 186.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4ubp.ent.gz pdb4ubp.ent.gz | 142.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4ubp.json.gz 4ubp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4ubp_validation.pdf.gz 4ubp_validation.pdf.gz | 453.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4ubp_full_validation.pdf.gz 4ubp_full_validation.pdf.gz | 467.4 KB | 表示 | |
| XML形式データ |  4ubp_validation.xml.gz 4ubp_validation.xml.gz | 38.5 KB | 表示 | |
| CIF形式データ |  4ubp_validation.cif.gz 4ubp_validation.cif.gz | 58.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ub/4ubp https://data.pdbj.org/pub/pdb/validation_reports/ub/4ubp ftp://data.pdbj.org/pub/pdb/validation_reports/ub/4ubp ftp://data.pdbj.org/pub/pdb/validation_reports/ub/4ubp | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2ubpS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-PROTEIN (UREASE (CHAIN ... , 3種, 3分子 ABC
| #1: タンパク質 | 分子量: 11187.017 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41022, urease Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41022, urease |
|---|---|
| #2: タンパク質 | 分子量: 13975.539 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41021, urease Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41021, urease |
| #3: タンパク質 | 分子量: 61646.766 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41020, urease Sporosarcina pasteurii (バクテリア) / 細胞内の位置: CYTOPLASM / 株: DSM 33 / 参照: UniProt: P41020, urease |
-非ポリマー , 3種, 749分子 
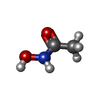



| #4: 化合物 | | #5: 化合物 | ChemComp-HAE / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.7 Å3/Da / 溶媒含有率: 54 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 6.3 詳細: ATED AMMONIUM SULPHATE, 1.2 M LICL), DROXAMIC ACID, 1OOMM SODIUM CITRATE PH 6.3. HANGING DROP 20 C, 3 UL PROTEIN SOLUTION (11 MG/ML IN 20 MM TRIS HCL PH 8.0 + 4MM ACETOHYDROXAMIC ACID) + 3 ...詳細: ATED AMMONIUM SULPHATE, 1.2 M LICL), DROXAMIC ACID, 1OOMM SODIUM CITRATE PH 6.3. HANGING DROP 20 C, 3 UL PROTEIN SOLUTION (11 MG/ML IN 20 MM TRIS HCL PH 8.0 + 4MM ACETOHYDROXAMIC ACID) + 3 MICROLITERS PRECIPITANT SOLUTION, pH 6.30 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶 | *PLUS 溶媒含有率: 54 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: BW7B / 波長: 0.8342 / ビームライン: BW7B / 波長: 0.8342 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1998年4月30日 / 詳細: BENT MIRROR |
| 放射 | モノクロメーター: SI(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.8342 Å / 相対比: 1 |
| 反射 | 解像度: 1.55→35 Å / Num. obs: 138830 / % possible obs: 99.5 % / Observed criterion σ(I): -3 / 冗長度: 26.02 % / Rmerge(I) obs: 0.071 / Rsym value: 0.071 / Net I/σ(I): 15.03 |
| 反射 シェル | 解像度: 1.55→1.58 Å / 冗長度: 4.29 % / Rmerge(I) obs: 0.479 / Mean I/σ(I) obs: 2.23 / Rsym value: 0.479 / % possible all: 99.9 |
| 反射 | *PLUS 最高解像度: 1.55 Å / 最低解像度: 32.5 Å / % possible obs: 99.5 % / Num. measured all: 3612270 |
| 反射 シェル | *PLUS 最高解像度: 1.55 Å / 最低解像度: 1.58 Å / % possible obs: 99.5 % / Mean I/σ(I) obs: 2.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 2UBP 解像度: 1.55→32.73 Å / 交差検証法: RFREE / σ(F): 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.22 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.55→32.73 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: REFMAC / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.151 / Rfactor Rwork: 0.151 / 最高解像度: 1.55 Å / 最低解像度: 32.7 Å / Rfactor Rfree: 0.19 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj



