+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4u6k | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of DNA/RNA duplex containing 2'-4'-BNA-NC | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA/RNA / DNA/RNA duplex / antisense / Bridged Nucleic Acid / DNA-RNA complex | ||||||
| 機能・相同性 | COBALT HEXAMMINE(III) / DNA / RNA 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 | synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.5 Å 多波長異常分散 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Kondo, J. / Nomura, Y. / Kitahara, Y. / Obika, S. / Torigoe, H. | ||||||
 引用 引用 |  ジャーナル: Chem.Commun.(Camb.) / 年: 2016 ジャーナル: Chem.Commun.(Camb.) / 年: 2016タイトル: The crystal structure of a 2',4'-BNA(NC)[N-Me]-modified antisense gapmer in complex with the target RNA. 著者: Kondo, J. / Nomura, Y. / Kitahara, Y. / Obika, S. / Torigoe, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4u6k.cif.gz 4u6k.cif.gz | 44.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4u6k.ent.gz pdb4u6k.ent.gz | 30.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4u6k.json.gz 4u6k.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4u6k_validation.pdf.gz 4u6k_validation.pdf.gz | 417.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4u6k_full_validation.pdf.gz 4u6k_full_validation.pdf.gz | 427.7 KB | 表示 | |
| XML形式データ |  4u6k_validation.xml.gz 4u6k_validation.xml.gz | 9.7 KB | 表示 | |
| CIF形式データ |  4u6k_validation.cif.gz 4u6k_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u6/4u6k https://data.pdbj.org/pub/pdb/validation_reports/u6/4u6k ftp://data.pdbj.org/pub/pdb/validation_reports/u6/4u6k ftp://data.pdbj.org/pub/pdb/validation_reports/u6/4u6k | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 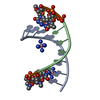
| ||||||||||||||||||||||||||||||
| 2 | 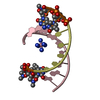
| ||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 2981.895 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) #2: DNA鎖 | 分子量: 2888.994 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) #3: 化合物 | #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 2 X線回折 / 使用した結晶の数: 2 |
|---|
- 試料調製
試料調製
| 結晶 |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 / 詳細: Na Cacodylate, NaCl, Hexammine Cobalt, MPD |
-データ収集
| 回折 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | 冗長度: 3.6 % / 数: 117593 / Rmerge(I) obs: 0.062 / Χ2: 0.98 / D res high: 1.6 Å / D res low: 51.21 Å / Num. obs: 32414 / % possible obs: 95.7 / Rejects: 882 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.5→25.61 Å / Num. obs: 21282 / % possible obs: 98 % / 冗長度: 6.82 % / Rmerge(I) obs: 0.056 / Χ2: 0.99 / Net I/σ(I): 17.1 / Num. measured all: 146347 / Scaling rejects: 1099 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
| 位相決定 | 手法:  多波長異常分散 多波長異常分散 | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Phasing dm | FOM : 0.64 / FOM acentric: 0.64 / FOM centric: 0.65 / 反射: 21207 / Reflection acentric: 19193 / Reflection centric: 2014 | ||||||||||||||||||||||||||||||||||||||||||
| Phasing dm shell |
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 1.5→25.61 Å / FOM work R set: 0.8754 / 交差検証法: FREE R-VALUE / σ(F): 0 多波長異常分散 / 解像度: 1.5→25.61 Å / FOM work R set: 0.8754 / 交差検証法: FREE R-VALUE / σ(F): 0
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 45.5582 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 82.04 Å2 / Biso mean: 22.3891 Å2 / Biso min: 8.48 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.5→25.61 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj