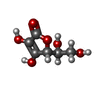
登録情報 データベース : PDB / ID : 4twlタイトル Crystal structure of dioscorin complexed with ascorbate Dioscorin 5 キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Dioscorea japonica (ジネンジョウ)手法 / / 解像度 : 2.11 Å データ登録者 Xue, Y.L. / Miyakawa, T. / Nakamura, A. / Tanokura, M. 資金援助 組織 認可番号 国 High Energy Accelerator Research Organization 2011G605 Targeted Proteins Research Program (TPRP) of the Ministry of Education, Culture, Sports, Science and Technology of Japan National Natural Science Foundation of China 31201285 Scientific Research Foundation for the Returned Overseas Chinese Scholars 2013693
ジャーナル : Mol Plant / 年 : 2015タイトル : Yam Tuber Storage Protein Reduces Plant Oxidants Using the Coupled Reactions as Carbonic Anhydrase and Dehydroascorbate Reductase著者 : Xue, Y.L. / Miyakawa, T. / Nakamura, A. / Hatano, K. / Sawano, Y. / Tanokura, M. 履歴 登録 2014年7月1日 登録サイト / 処理サイト 改定 1.0 2015年4月1日 Provider / タイプ 改定 1.1 2015年8月5日 Group 改定 1.2 2020年1月29日 Group / Derived calculations / カテゴリ / pdbx_struct_oper_listItem / _pdbx_struct_oper_list.symmetry_operation改定 1.3 2020年7月29日 Group / Derived calculations / カテゴリ / struct_site / struct_site_gen / Item / 解説 / Provider / タイプ 改定 1.4 2024年10月30日 Group / Database references / Structure summaryカテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Dioscorea japonica (ジネンジョウ)
Dioscorea japonica (ジネンジョウ) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.11 Å
シンクロトロン / 解像度: 2.11 Å  データ登録者
データ登録者 日本,
日本,  中国, 4件
中国, 4件  引用
引用 ジャーナル: Mol Plant / 年: 2015
ジャーナル: Mol Plant / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4twl.cif.gz
4twl.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4twl.ent.gz
pdb4twl.ent.gz PDB形式
PDB形式 4twl.json.gz
4twl.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4twl_validation.pdf.gz
4twl_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4twl_full_validation.pdf.gz
4twl_full_validation.pdf.gz 4twl_validation.xml.gz
4twl_validation.xml.gz 4twl_validation.cif.gz
4twl_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/tw/4twl
https://data.pdbj.org/pub/pdb/validation_reports/tw/4twl ftp://data.pdbj.org/pub/pdb/validation_reports/tw/4twl
ftp://data.pdbj.org/pub/pdb/validation_reports/tw/4twl リンク
リンク 集合体
集合体


 要素
要素 Dioscorea japonica (ジネンジョウ)
Dioscorea japonica (ジネンジョウ)
 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Photon Factory
Photon Factory  / ビームライン: BL-5A / 波長: 1 Å
/ ビームライン: BL-5A / 波長: 1 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj