| 登録情報 | データベース: PDB / ID: 4tot
|
|---|
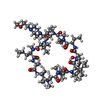
| タイトル | Crystal structure of rat cyclophilin D in complex with a potent nonimmunosuppressive inhibitor |
|---|
 要素 要素 | - Peptidyl-prolyl cis-trans isomerase F, mitochondrial
- nonimmunosuppressive inhibitor
|
|---|
 キーワード キーワード | ISOMERASE/ISOMERASE Inhibitor / Inhibitor / Complex / Cyclosporin / ISOMERASE-ISOMERASE Inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
: / regulation of proton-transporting ATPase activity, rotational mechanism / negative regulation of oxidative phosphorylation uncoupler activity / mitochondrial outer membrane permeabilization involved in programmed cell death / regulation of mitochondrial membrane permeability involved in programmed necrotic cell death / mitochondrial permeability transition pore complex / cellular response to arsenic-containing substance / negative regulation of ATP-dependent activity / negative regulation of oxidative phosphorylation / regulation of mitochondrial membrane permeability ...: / regulation of proton-transporting ATPase activity, rotational mechanism / negative regulation of oxidative phosphorylation uncoupler activity / mitochondrial outer membrane permeabilization involved in programmed cell death / regulation of mitochondrial membrane permeability involved in programmed necrotic cell death / mitochondrial permeability transition pore complex / cellular response to arsenic-containing substance / negative regulation of ATP-dependent activity / negative regulation of oxidative phosphorylation / regulation of mitochondrial membrane permeability / cyclosporin A binding / negative regulation of release of cytochrome c from mitochondria / necroptotic process / positive regulation of release of cytochrome c from mitochondria / apoptotic mitochondrial changes / protein peptidyl-prolyl isomerization / negative regulation of intrinsic apoptotic signaling pathway / cellular response to calcium ion / peptidylprolyl isomerase / peptidyl-prolyl cis-trans isomerase activity / response to ischemia / peptide binding / cellular response to hydrogen peroxide / protein folding / regulation of apoptotic process / response to oxidative stress / mitochondrial inner membrane / mitochondrial matrix / intracellular membrane-bounded organelle / negative regulation of apoptotic process / mitochondrion / cytoplasm類似検索 - 分子機能 Cyclophilin-like / Cyclophilin / Cyclophilin-type peptidyl-prolyl cis-trans isomerase, conserved site / Cyclophilin-type peptidyl-prolyl cis-trans isomerase signature. / Cyclophilin-type peptidyl-prolyl cis-trans isomerase domain profile. / Cyclophilin-type peptidyl-prolyl cis-trans isomerase domain / Cyclophilin type peptidyl-prolyl cis-trans isomerase/CLD / Cyclophilin-like domain superfamily / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 nonimmunosuppressive inhibitor / Peptidyl-prolyl cis-trans isomerase F, mitochondrial類似検索 - 構成要素 |
|---|
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.39 Å 分子置換 / 解像度: 2.39 Å |
|---|
 データ登録者 データ登録者 | Knapp, M.S. / Elling, R.A. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2014 ジャーナル: J.Med.Chem. / 年: 2014
タイトル: Potent nonimmunosuppressive cyclophilin inhibitors with improved pharmaceutical properties and decreased transporter inhibition.
著者: Fu, J. / Tjandra, M. / Becker, C. / Bednarczyk, D. / Capparelli, M. / Elling, R. / Hanna, I. / Fujimoto, R. / Furegati, M. / Karur, S. / Kasprzyk, T. / Knapp, M. / Leung, K. / Li, X. / Lu, P. ...著者: Fu, J. / Tjandra, M. / Becker, C. / Bednarczyk, D. / Capparelli, M. / Elling, R. / Hanna, I. / Fujimoto, R. / Furegati, M. / Karur, S. / Kasprzyk, T. / Knapp, M. / Leung, K. / Li, X. / Lu, P. / Mergo, W. / Miault, C. / Ng, S. / Parker, D. / Peng, Y. / Roggo, S. / Rivkin, A. / Simmons, R.L. / Wang, M. / Wiedmann, B. / Weiss, A.H. / Xiao, L. / Xie, L. / Xu, W. / Yifru, A. / Yang, S. / Zhou, B. / Sweeney, Z.K. |
|---|
| 履歴 | | 登録 | 2014年6月6日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年11月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年12月27日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other / Refinement description / Source and taxonomy
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / citation / database_2 / entity_src_gen / pdbx_database_status / pdbx_entity_src_syn / pdbx_struct_oper_list / refine_hist / struct_conn / struct_ref_seq
Item: _citation.journal_id_CSD / _database_2.pdbx_DOI ..._citation.journal_id_CSD / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _entity_src_gen.pdbx_alt_source_flag / _pdbx_database_status.pdb_format_compatible / _pdbx_entity_src_syn.pdbx_alt_source_flag / _pdbx_struct_oper_list.symmetry_operation / _refine_hist.number_atoms_solvent / _refine_hist.number_atoms_total / _refine_hist.pdbx_number_atoms_nucleic_acid / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end |
|---|
| 改定 1.2 | 2024年4月3日 | Group: Refinement description / カテゴリ: pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 2.39 Å
分子置換 / 解像度: 2.39 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2014
ジャーナル: J.Med.Chem. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4tot.cif.gz
4tot.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4tot.ent.gz
pdb4tot.ent.gz PDB形式
PDB形式 4tot.json.gz
4tot.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/to/4tot
https://data.pdbj.org/pub/pdb/validation_reports/to/4tot ftp://data.pdbj.org/pub/pdb/validation_reports/to/4tot
ftp://data.pdbj.org/pub/pdb/validation_reports/to/4tot リンク
リンク 集合体
集合体
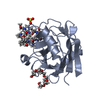



 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E+ SUPERBRIGHT / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E+ SUPERBRIGHT / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












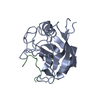
 PDBj
PDBj