+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4qtk | ||||||
|---|---|---|---|---|---|---|---|
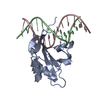
| タイトル | Complex of WOPR domain of Wor1 in Candida albicans with the 17bp dsDNA | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSCRIPTION/DNA / Wor1 / WOPR domain / white-opaque switching / protein-DNA recognition / transcriptional regulation / DNA binding domain / TRANSCRIPTION-DNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of phenotypic switching / parasexual reproduction with cellular fusion / regulation of single-species biofilm formation on inanimate substrate / regulation of phenotypic switching / biological process involved in symbiotic interaction / filamentous growth of a population of unicellular organisms / mating / single-species biofilm formation on inanimate substrate / phenotypic switching / adhesion of symbiont to host ...positive regulation of phenotypic switching / parasexual reproduction with cellular fusion / regulation of single-species biofilm formation on inanimate substrate / regulation of phenotypic switching / biological process involved in symbiotic interaction / filamentous growth of a population of unicellular organisms / mating / single-species biofilm formation on inanimate substrate / phenotypic switching / adhesion of symbiont to host / sequence-specific DNA binding / cell adhesion / DNA-binding transcription factor activity / regulation of DNA-templated transcription / chromatin / positive regulation of transcription by RNA polymerase II / DNA binding / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Candida albicans SC5314 (酵母) Candida albicans SC5314 (酵母) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2.99 Å 単波長異常分散 / 解像度: 2.99 Å | ||||||
 データ登録者 データ登録者 | Zhang, S. / Zhang, T. / Ding, J. | ||||||
 引用 引用 |  ジャーナル: Cell Res. / 年: 2014 ジャーナル: Cell Res. / 年: 2014タイトル: Crystal structure of the WOPR-DNA complex and implications for Wor1 function in white-opaque switching of Candida albicans. 著者: Zhang, S. / Zhang, T. / Yan, M. / Ding, J. / Chen, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4qtk.cif.gz 4qtk.cif.gz | 112.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4qtk.ent.gz pdb4qtk.ent.gz | 81.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4qtk.json.gz 4qtk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4qtk_validation.pdf.gz 4qtk_validation.pdf.gz | 461.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4qtk_full_validation.pdf.gz 4qtk_full_validation.pdf.gz | 468.2 KB | 表示 | |
| XML形式データ |  4qtk_validation.xml.gz 4qtk_validation.xml.gz | 14.7 KB | 表示 | |
| CIF形式データ |  4qtk_validation.cif.gz 4qtk_validation.cif.gz | 19.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qt/4qtk https://data.pdbj.org/pub/pdb/validation_reports/qt/4qtk ftp://data.pdbj.org/pub/pdb/validation_reports/qt/4qtk ftp://data.pdbj.org/pub/pdb/validation_reports/qt/4qtk | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 30818.729 Da / 分子数: 2 / 断片: UNP RESIDUES 6-272 / 由来タイプ: 組換発現 / 由来: (組換発現)  Candida albicans SC5314 (酵母) / 遺伝子: WOR1, EAP2, TOS9, CaO19.12348, CaO19.4884 / プラスミド: pETDuet / 発現宿主: Candida albicans SC5314 (酵母) / 遺伝子: WOR1, EAP2, TOS9, CaO19.12348, CaO19.4884 / プラスミド: pETDuet / 発現宿主:  #2: DNA鎖 | 分子量: 5193.423 Da / 分子数: 2 / 由来タイプ: 合成 #3: DNA鎖 | 分子量: 5215.419 Da / 分子数: 2 / 由来タイプ: 合成 #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.91 Å3/Da / 溶媒含有率: 35.71 % / Mosaicity: 1.254 ° |
|---|---|
| 結晶化 | 温度: 289 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.7 詳細: 0.10M Bis-Tris, 0.2M ammonnuim sulfate, 20% PEG 3350, pH 5.7, temperature 289K, VAPOR DIFFUSION, HANGING DROP |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL17U / 波長: 0.9793 Å / ビームライン: BL17U / 波長: 0.9793 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2012年11月29日 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 0.9793 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.99→50 Å / Num. obs: 12474 / % possible obs: 97.7 % / 冗長度: 7.2 % / Rmerge(I) obs: 0.107 / Χ2: 1.943 / Net I/σ(I): 15.7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: _
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単波長異常分散 / 解像度: 2.99→38.86 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.911 / SU B: 22.974 / SU ML: 0.415 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.469 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 単波長異常分散 / 解像度: 2.99→38.86 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.911 / SU B: 22.974 / SU ML: 0.415 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.469 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 207.4 Å2 / Biso mean: 84.634 Å2 / Biso min: 40.46 Å2
| ||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.99→38.86 Å
| ||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.994→3.072 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







































