| 登録情報 | データベース: PDB / ID: 4pqo
|
|---|
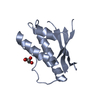
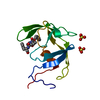
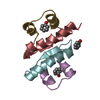
| タイトル | Structure of the human SNX14 PX domain in space group I41 |
|---|
 要素 要素 | Sorting nexin-14 |
|---|
 キーワード キーワード | PROTEIN TRANSPORT / sorting nexin / phox homology domain / phosphoinositide binding |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
phosphatidylinositol-3,5-bisphosphate binding / autophagosome maturation / postsynaptic modulation of chemical synaptic transmission / phosphatidylinositol binding / late endosome membrane / late endosome / protein transport / lysosome / postsynapse / lysosomal membrane ...phosphatidylinositol-3,5-bisphosphate binding / autophagosome maturation / postsynaptic modulation of chemical synaptic transmission / phosphatidylinositol binding / late endosome membrane / late endosome / protein transport / lysosome / postsynapse / lysosomal membrane / intracellular membrane-bounded organelle / dendrite / cytosol類似検索 - 分子機能 Sorting nexin-14, PX domain / SNX14, RGS domain / Phox-associated domain / Sorting nexin, C-terminal / PXA domain / Sorting nexin C terminal / PXA domain profile. / Domain associated with PX domains / Phox-like domain / PX Domain ...Sorting nexin-14, PX domain / SNX14, RGS domain / Phox-associated domain / Sorting nexin, C-terminal / PXA domain / Sorting nexin C terminal / PXA domain profile. / Domain associated with PX domains / Phox-like domain / PX Domain / PhoX homologous domain, present in p47phox and p40phox. / PX domain profile. / PX domain / Phox homology / PX domain superfamily / Regulator of G protein signaling domain / RGS domain / RGS domain profile. / Regulator of G protein signalling domain / RGS, subdomain 2 / RGS domain superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.55 Å 分子置換 / 解像度: 2.55 Å |
|---|
 データ登録者 データ登録者 | Mas, C. / Norwood, S. / Bugarcic, A. / Kinna, G. / Leneva, N. / Kovtun, O. / Teasdale, R. / Collins, B. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2014 ジャーナル: J.Biol.Chem. / 年: 2014
タイトル: Structural Basis for Different Phosphoinositide Specificities of the PX Domains of Sorting Nexins Regulating G-protein Signaling.
著者: Mas, C. / Norwood, S.J. / Bugarcic, A. / Kinna, G. / Leneva, N. / Kovtun, O. / Ghai, R. / Ona Yanez, L.E. / Davis, J.L. / Teasdale, R.D. / Collins, B.M. |
|---|
| 履歴 | | 登録 | 2014年3月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年9月3日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年10月29日 | Group: Database references |
|---|
| 改定 1.2 | 2024年2月28日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 2.55 Å
分子置換 / 解像度: 2.55 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2014
ジャーナル: J.Biol.Chem. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4pqo.cif.gz
4pqo.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4pqo.ent.gz
pdb4pqo.ent.gz PDB形式
PDB形式 4pqo.json.gz
4pqo.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4pqo_validation.pdf.gz
4pqo_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4pqo_full_validation.pdf.gz
4pqo_full_validation.pdf.gz 4pqo_validation.xml.gz
4pqo_validation.xml.gz 4pqo_validation.cif.gz
4pqo_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/pq/4pqo
https://data.pdbj.org/pub/pdb/validation_reports/pq/4pqo ftp://data.pdbj.org/pub/pdb/validation_reports/pq/4pqo
ftp://data.pdbj.org/pub/pdb/validation_reports/pq/4pqo リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: SNX14 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SNX14 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å 解析
解析 分子置換 / 解像度: 2.55→22.098 Å / σ(F): 1.93 / 位相誤差: 23.12 / 立体化学のターゲット値: TWIN_LSQ_F
分子置換 / 解像度: 2.55→22.098 Å / σ(F): 1.93 / 位相誤差: 23.12 / 立体化学のターゲット値: TWIN_LSQ_F ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

