[English] 日本語
 Yorodumi
Yorodumi- PDB-4nmc: Crystal structure of oxidized proline utilization A (PutA) from G... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4nmc | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of oxidized proline utilization A (PutA) from Geobacter sulfurreducens PCA complexed with Zwittergent 3-12 | ||||||
 Components Components | Proline dehydrogenase and Delta-1-pyrroline-5-carboxylate dehydrogenase | ||||||
 Keywords Keywords | OXIDOREDUCTASE / flavoenzyme / Rossmann fold / aldehyde dehydrogenase / flavin adenine dinucleotide / nicotinamide adenine dinucleotide / proline catabolism / substrate channeling / bifunctional enzyme | ||||||
| Function / homology |  Function and homology information Function and homology informationproline dehydrogenase activity / L-glutamate gamma-semialdehyde dehydrogenase / L-glutamate gamma-semialdehyde dehydrogenase activity / L-proline catabolic process to L-glutamate / cytoplasmic side of plasma membrane / DNA-binding transcription factor activity / nucleotide binding Similarity search - Function | ||||||
| Biological species |  Geobacter sulfurreducens (bacteria) Geobacter sulfurreducens (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  SAD / Resolution: 1.901 Å SAD / Resolution: 1.901 Å | ||||||
 Authors Authors | Singh, H. / Tanner, J.J. | ||||||
 Citation Citation |  Journal: Proc.Natl.Acad.Sci.USA / Year: 2014 Journal: Proc.Natl.Acad.Sci.USA / Year: 2014Title: Structures of the PutA peripheral membrane flavoenzyme reveal a dynamic substrate-channeling tunnel and the quinone-binding site. Authors: Singh, H. / Arentson, B.W. / Becker, D.F. / Tanner, J.J. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4nmc.cif.gz 4nmc.cif.gz | 764 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4nmc.ent.gz pdb4nmc.ent.gz | 620.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4nmc.json.gz 4nmc.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  4nmc_validation.pdf.gz 4nmc_validation.pdf.gz | 1.2 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  4nmc_full_validation.pdf.gz 4nmc_full_validation.pdf.gz | 1.2 MB | Display | |
| Data in XML |  4nmc_validation.xml.gz 4nmc_validation.xml.gz | 75.1 KB | Display | |
| Data in CIF |  4nmc_validation.cif.gz 4nmc_validation.cif.gz | 109.8 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/nm/4nmc https://data.pdbj.org/pub/pdb/validation_reports/nm/4nmc ftp://data.pdbj.org/pub/pdb/validation_reports/nm/4nmc ftp://data.pdbj.org/pub/pdb/validation_reports/nm/4nmc | HTTPS FTP |
-Related structure data
| Related structure data |  4nm9C  4nmaC  4nmbC  4nmdC  4nmeC  4nmfC C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 112324.672 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Geobacter sulfurreducens (bacteria) / Strain: ATCC 51573 / DSM 12127 / PCA / Gene: putA, GSU3395 / Plasmid: pNIC28-Bsa4 / Production host: Geobacter sulfurreducens (bacteria) / Strain: ATCC 51573 / DSM 12127 / PCA / Gene: putA, GSU3395 / Plasmid: pNIC28-Bsa4 / Production host:  References: UniProt: Q746X3, EC: 1.5.99.8, EC: 1.5.1.12, L-glutamate gamma-semialdehyde dehydrogenase |
|---|
-Non-polymers , 6 types, 1131 molecules 


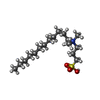
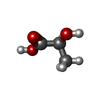






| #2: Chemical | | #3: Chemical | #4: Chemical | #5: Chemical | ChemComp-C15 / | #6: Chemical | ChemComp-2OP / ( | #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.14 Å3/Da / Density % sol: 60.82 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: microbatch Details: 0.10-0.25 M potassium sodium tartrate, 20-25% w/v PEG3350, MICROBATCH, temperature 298K |
-Data collection
| Diffraction | Mean temperature: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 4.2.2 / Wavelength: 1 Å / Beamline: 4.2.2 / Wavelength: 1 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: NOIR-1 / Detector: CCD / Date: Apr 7, 2012 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: Rosenbaum-Rock double crystal Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Redundancy: 4.1 % / Av σ(I) over netI: 7.1 / Number: 369061 / Rsym value: 0.105 / D res high: 2.568 Å / D res low: 121.065 Å / Num. obs: 90248 / % possible obs: 99.8 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.891→47.039 Å / Num. all: 220275 / Num. obs: 220275 / % possible obs: 99.4 % / Redundancy: 5.3 % / Rpim(I) all: 0.046 / Rrim(I) all: 0.109 / Rsym value: 0.098 / Net I/av σ(I): 6.511 / Net I/σ(I): 10.4 / Num. measured all: 1166275 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  SAD / Resolution: 1.901→47.039 Å / Occupancy max: 1 / Occupancy min: 0.37 / FOM work R set: 0.8695 / SU ML: 0.2 / σ(F): 0 / Phase error: 20.53 / Stereochemistry target values: ML SAD / Resolution: 1.901→47.039 Å / Occupancy max: 1 / Occupancy min: 0.37 / FOM work R set: 0.8695 / SU ML: 0.2 / σ(F): 0 / Phase error: 20.53 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.2 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 105.06 Å2 / Biso mean: 31.0247 Å2 / Biso min: 8.35 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.901→47.039 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 30
|
 Movie
Movie Controller
Controller












 PDBj
PDBj







