| 登録情報 | データベース: PDB / ID: 4h82
|
|---|
| タイトル | Crystal structure of mutant MMP-9 catalytic domain in complex with a twin inhibitor. |
|---|
 要素 要素 | Matrix metalloproteinase-9 |
|---|
 キーワード キーワード | hydrolase/hydrolase inhibitor / HYDROLASE/TWIN INHIBITOR / Zincin-like / Gelatinase / Collagenase (Catalytic Domain) / hydrolase-hydrolase inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
gelatinase B / negative regulation of epithelial cell differentiation involved in kidney development / negative regulation of cation channel activity / negative regulation of cysteine-type endopeptidase activity involved in apoptotic signaling pathway / cellular response to UV-A / positive regulation of keratinocyte migration / regulation of neuroinflammatory response / positive regulation of epidermal growth factor receptor signaling pathway / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases ...gelatinase B / negative regulation of epithelial cell differentiation involved in kidney development / negative regulation of cation channel activity / negative regulation of cysteine-type endopeptidase activity involved in apoptotic signaling pathway / cellular response to UV-A / positive regulation of keratinocyte migration / regulation of neuroinflammatory response / positive regulation of epidermal growth factor receptor signaling pathway / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases / endodermal cell differentiation / positive regulation of release of cytochrome c from mitochondria / response to amyloid-beta / macrophage differentiation / Collagen degradation / collagen catabolic process / EPH-ephrin mediated repulsion of cells / extracellular matrix disassembly / ephrin receptor signaling pathway / positive regulation of DNA binding / negative regulation of intrinsic apoptotic signaling pathway / positive regulation of vascular associated smooth muscle cell proliferation / collagen binding / cellular response to cadmium ion / embryo implantation / Degradation of the extracellular matrix / extracellular matrix organization / positive regulation of receptor binding / skeletal system development / Signaling by SCF-KIT / metalloendopeptidase activity / cellular response to reactive oxygen species / metallopeptidase activity / cell migration / tertiary granule lumen / peptidase activity / cellular response to lipopolysaccharide / Interleukin-4 and Interleukin-13 signaling / collagen-containing extracellular matrix / endopeptidase activity / ficolin-1-rich granule lumen / Extra-nuclear estrogen signaling / positive regulation of protein phosphorylation / positive regulation of apoptotic process / serine-type endopeptidase activity / apoptotic process / Neutrophil degranulation / negative regulation of apoptotic process / proteolysis / extracellular space / zinc ion binding / extracellular exosome / extracellular region / identical protein binding類似検索 - 分子機能 Fibronectin type II domain / Fibronectin type II domain superfamily / Fibronectin type II domain / Fibronectin type-II collagen-binding domain signature. / Fibronectin type-II collagen-binding domain profile. / Fibronectin type 2 domain / Hemopexin, conserved site / Hemopexin domain signature. / Hemopexin-like domain / Peptidase M10A, cysteine switch, zinc binding site ...Fibronectin type II domain / Fibronectin type II domain superfamily / Fibronectin type II domain / Fibronectin type-II collagen-binding domain signature. / Fibronectin type-II collagen-binding domain profile. / Fibronectin type 2 domain / Hemopexin, conserved site / Hemopexin domain signature. / Hemopexin-like domain / Peptidase M10A, cysteine switch, zinc binding site / Matrixins cysteine switch. / Hemopexin-like repeats / Hemopexin-like domain superfamily / Hemopexin / Hemopexin repeat profile. / Hemopexin-like repeats. / Peptidase M10A / Peptidase M10A, catalytic domain / Peptidase M10, metallopeptidase / Matrixin / PGBD-like superfamily / Peptidase, metallopeptidase / Zinc-dependent metalloprotease / Collagenase (Catalytic Domain) / Collagenase (Catalytic Domain) / Metallopeptidase, catalytic domain superfamily / Kringle-like fold / Neutral zinc metallopeptidases, zinc-binding region signature. / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Chem-L29 / DI(HYDROXYETHYL)ETHER / S-1,2-PROPANEDIOL / PROPANOIC ACID / Matrix metalloproteinase-9類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Antoni, C. / Stura, E.A. / Vera, L. / Cassar-Lajeunesse, E. / Nuti, E. / Dive, V. / Rossello, A. |
|---|
 引用 引用 |  ジャーナル: J.Struct.Biol. / 年: 2013タイトル ジャーナル: J.Struct.Biol. / 年: 2013タイトル: Crystallization of bi-functional ligand protein complexes. 著者: Antoni, C. / Vera, L. / Devel, L. / Catalani, M.P. / Czarny, B. / Cassar-Lajeunesse, E. / Nuti, E. / Rossello, A. / Dive, V. / Stura, E.A.#1: ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2005タイトル: A new development of matrix metalloproteinase inhibitors: twin hydroxamic acids as potent inhibitors of MMPs. 著者: Rossello, A. / Nuti, E. / Catalani, M.P. / Carelli, P. / Orlandini, E. / Rapposelli, S. / Tuccinardi, T. / Atkinson, S.J. / Murphy, G. / Balsamo, A. |
|---|
| 履歴 | | 登録 | 2012年9月21日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2013年5月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年10月8日 | Group: Database references |
|---|
| 改定 1.2 | 2014年10月15日 | Group: Derived calculations |
|---|
| 改定 1.3 | 2015年8月12日 | Group: Database references |
|---|
| 改定 1.4 | 2017年8月9日 | Group: Refinement description / Source and taxonomy / カテゴリ: entity_src_gen / software |
|---|
| 改定 1.5 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Struct.Biol. / 年: 2013
ジャーナル: J.Struct.Biol. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4h82.cif.gz
4h82.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4h82.ent.gz
pdb4h82.ent.gz PDB形式
PDB形式 4h82.json.gz
4h82.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4h82_validation.pdf.gz
4h82_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4h82_full_validation.pdf.gz
4h82_full_validation.pdf.gz 4h82_validation.xml.gz
4h82_validation.xml.gz 4h82_validation.cif.gz
4h82_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/h8/4h82
https://data.pdbj.org/pub/pdb/validation_reports/h8/4h82 ftp://data.pdbj.org/pub/pdb/validation_reports/h8/4h82
ftp://data.pdbj.org/pub/pdb/validation_reports/h8/4h82 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: CLG4B, MMP9 / プラスミド: pET-14b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CLG4B, MMP9 / プラスミド: pET-14b / 発現宿主: 



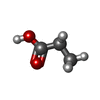

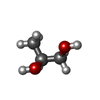








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.98011
/ ビームライン: PROXIMA 1 / 波長: 0.98011  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj


















