回折 ID 平均測定温度 (K)Crystal-ID 1 100 1 2 100 2
放射光源 由来 / タイプ / 波長 検出器 タイプ ID 検出器 日付 1 2011年9月6日 2 2011年9月15日
放射 ID モノクロメーター プロトコル 単色(M)・ラウエ(L) 散乱光タイプ Wavelength-ID 1 OSMIC VARIMAXSINGLE WAVELENGTHMx-ray 1 2 OSMIC VARIMAXSINGLE WAVELENGTHMx-ray 1
放射波長 波長 / 相対比 Reflection 冗長度 / Av σ(I) over netI / 数 / Rmerge (I) obs / Χ2 / D res high / D res low / Num. obs / % possible obs Diffraction reflection shell 大きな表を表示 (7 x 20) 大きな表を隠す 最高解像度 (Å)最低解像度 (Å)% possible obs (%)ID Rmerge (I) obs Chi squared Redundancy 7.13 50 99.8 1 0.047 0.913 13.6 5.66 7.13 100 1 0.076 1.098 14.8 4.95 5.66 100 1 0.107 1.057 15.3 4.5 4.95 100 1 0.132 1.165 15.2 4.17 4.5 100 1 0.146 1.017 15.6 3.93 4.17 100 1 0.174 1.066 15.4 3.73 3.93 100 1 0.227 1.081 15.9 3.57 3.73 100 1 0.242 1.028 16 3.43 3.57 100 1 0.37 1.081 15.3 3.31 3.43 99.2 1 0.229 1.092 7.2 3.21 3.31 98.1 1 0.131 1.04 6.1 3.12 3.21 98.9 1 0.194 1.077 6 3.04 3.12 98.1 1 0.381 1.059 5.9 2.96 3.04 99.2 1 0.528 1.092 5.9 2.89 2.96 99.2 1 0.628 1.083 6.1 2.83 2.89 99.2 1 0.629 1.07 5.8 2.78 2.83 99.2 1 0.662 1.094 6 2.72 2.78 100 1 0.736 1.084 5.7 2.68 2.72 99.7 1 0.541 1.075 5.9 2.63 2.68 99.5 1 0.447 1.033 5.9
反射 解像度 / Num. all / Num. obs / % possible obs / Observed criterion σ(F) / Observed criterion σ(I) / 冗長度 / Rmerge (I) obs / Χ2 / Net I/σ(I) 反射 シェル 大きな表を表示 (7 x 20) 大きな表を隠す 解像度 (Å)冗長度 (%)Rmerge (I) obs Num. unique all Χ2 Diffraction-ID % possible all 2.64-2.68 1.8 0.188 344 0.923 1,295 2.68-2.73 1.8 0.248 368 0.883 1,299.5 2.73-2.79 1.8 0.305 348 0.84 1,299.4 2.79-2.84 1.8 0.232 379 0.919 1,299 2.84-2.9 1.7 0.281 349 0.913 1,299.1 2.9-2.97 1.8 0.265 370 0.968 1,298.9 2.97-3.05 1.8 0.208 352 0.992 1,299.2 3.05-3.13 1.7 0.149 369 0.887 1,298.9 3.13-3.22 1.8 0.068 364 0.957 1,298.6 3.22-3.32 1.7 0.043 368 0.975 1,298.7 3.32-3.44 1.7 0.035 357 0.795 1,298.1 3.44-3.58 3.3 0.234 387 3.221 1,299.2 3.58-3.74 3.5 0.094 364 1.38 1,299.5 3.74-3.94 3.4 0.092 377 1.375 1,299.2 3.94-4.19 3.3 0.08 384 1.72 1,299 4.19-4.51 3.3 0.068 378 1.884 1,299.2 4.51-4.97 3.3 0.065 393 2.398 1,298.7 4.97-5.68 3.2 0.059 398 2.943 1,298.8 5.68-7.16 3.1 0.051 412 3.771 1,298.3 7.16-99 2.8 0.108 456 20.035 1,294
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  単一同系置換・異常分散 / 解像度: 2.64 Å
単一同系置換・異常分散 / 解像度: 2.64 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2013
ジャーナル: J.Biol.Chem. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4f2j.cif.gz
4f2j.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4f2j.ent.gz
pdb4f2j.ent.gz PDB形式
PDB形式 4f2j.json.gz
4f2j.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/f2/4f2j
https://data.pdbj.org/pub/pdb/validation_reports/f2/4f2j ftp://data.pdbj.org/pub/pdb/validation_reports/f2/4f2j
ftp://data.pdbj.org/pub/pdb/validation_reports/f2/4f2j リンク
リンク 集合体
集合体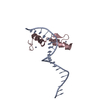
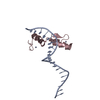
 要素
要素 Homo sapiens (ヒト) / 遺伝子: ZNF217, ZABC1 / プラスミド: pMAL / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ZNF217, ZABC1 / プラスミド: pMAL / 発現宿主: 
 X線回折 / 使用した結晶の数: 2
X線回折 / 使用した結晶の数: 2  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.5418 Å 単一同系置換・異常分散
単一同系置換・異常分散 解析
解析 単一同系置換・異常分散 / 解像度: 2.64→48.31 Å / Cor.coef. Fo:Fc: 0.924 / Cor.coef. Fo:Fc free: 0.969 / WRfactor Rfree: 0.2778 / WRfactor Rwork: 0.2563 / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7219 / SU B: 32.121 / SU ML: 0.285 / SU R Cruickshank DPI: 0.3269 / SU Rfree: 0.2467 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.327 / ESU R Free: 0.247 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
単一同系置換・異常分散 / 解像度: 2.64→48.31 Å / Cor.coef. Fo:Fc: 0.924 / Cor.coef. Fo:Fc free: 0.969 / WRfactor Rfree: 0.2778 / WRfactor Rwork: 0.2563 / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7219 / SU B: 32.121 / SU ML: 0.285 / SU R Cruickshank DPI: 0.3269 / SU Rfree: 0.2467 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.327 / ESU R Free: 0.247 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj









































