+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bt0 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | MuB is an AAAplus ATPase that forms helical filaments to control target selection for DNA transposition | ||||||
 要素 要素 | (TRANSCRIPTIONAL REGULATOR) x 2 | ||||||
 キーワード キーワード | TRANSCRIPTION / AAA+ ATPASE / DNA TRANSPOSITION / NUCLEOPROTEIN FILAMENT / SYMMETRY MISMATCH | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA-binding transcription activator activity / phosphorelay signal transduction system / cis-regulatory region sequence-specific DNA binding / protein-DNA complex / positive regulation of DNA-templated transcription / ATP hydrolysis activity / ATP binding / metal ion binding / identical protein binding 類似検索 - 分子機能 | ||||||
| 生物種 |  ENTEROBACTERIA PHAGE MU (ファージ) ENTEROBACTERIA PHAGE MU (ファージ) | ||||||
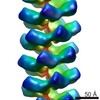
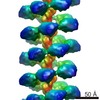
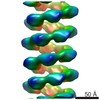
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 17 Å | ||||||
 データ登録者 データ登録者 | Mizuno, N. / Dramicanin, M. / Mizuuchi, M. / Adam, J. / Wang, Y. / Han, Y.W. / Yang, W. / Steven, A.C. / Mizuuchi, K. / Ramon-Maiques, S. | ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2013 ジャーナル: Proc Natl Acad Sci U S A / 年: 2013タイトル: MuB is an AAA+ ATPase that forms helical filaments to control target selection for DNA transposition. 著者: Naoko Mizuno / Marija Dramićanin / Michiyo Mizuuchi / Julia Adam / Yi Wang / Yong-Woon Han / Wei Yang / Alasdair C Steven / Kiyoshi Mizuuchi / Santiago Ramón-Maiques /  要旨: MuB is an ATP-dependent nonspecific DNA-binding protein that regulates the activity of the MuA transposase and captures target DNA for transposition. Mechanistic understanding of MuB function has ...MuB is an ATP-dependent nonspecific DNA-binding protein that regulates the activity of the MuA transposase and captures target DNA for transposition. Mechanistic understanding of MuB function has previously been hindered by MuB's poor solubility. Here we combine bioinformatic, mutagenic, biochemical, and electron microscopic analyses to unmask the structure and function of MuB. We demonstrate that MuB is an ATPase associated with diverse cellular activities (AAA+ ATPase) and forms ATP-dependent filaments with or without DNA. We also identify critical residues for MuB's ATPase, DNA binding, protein polymerization, and MuA interaction activities. Using single-particle electron microscopy, we show that MuB assembles into a helical filament, which binds the DNA in the axial channel. The helical parameters of the MuB filament do not match those of the coated DNA. Despite this protein-DNA symmetry mismatch, MuB does not deform the DNA duplex. These findings, together with the influence of MuB filament size on strand-transfer efficiency, lead to a model in which MuB-imposed symmetry transiently deforms the DNA at the boundary of the MuB filament and results in a bent DNA favored by MuA for transposition. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bt0.cif.gz 4bt0.cif.gz | 63.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bt0.ent.gz pdb4bt0.ent.gz | 45.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bt0.json.gz 4bt0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bt/4bt0 https://data.pdbj.org/pub/pdb/validation_reports/bt/4bt0 ftp://data.pdbj.org/pub/pdb/validation_reports/bt/4bt0 ftp://data.pdbj.org/pub/pdb/validation_reports/bt/4bt0 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | x 7
| ||||||||||||||||||||||||||||||||
| 2 |
| ||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||
| 対称性 | らせん対称: (回転対称性: 1 / Dyad axis: no / N subunits divisor: 1 / Num. of operations: 7 / Rise per n subunits: 9.01 Å / Rotation per n subunits: 66.2 °) | ||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 8562.894 Da / 分子数: 1 / 断片: AAAPLUS DOMAIN, RESIDUES 312-384 / 由来タイプ: 組換発現 由来: (組換発現)  ENTEROBACTERIA PHAGE MU (ファージ) ENTEROBACTERIA PHAGE MU (ファージ)発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 19397.400 Da / 分子数: 1 / 断片: AAAPLUS DOMAIN, RESIDUES 137-309 / 由来タイプ: 組換発現 由来: (組換発現)  ENTEROBACTERIA PHAGE MU (ファージ) ENTEROBACTERIA PHAGE MU (ファージ)発現宿主:  |
| #3: 化合物 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: MUB FILAMENT WITH DNA / タイプ: COMPLEX |
|---|---|
| 緩衝液 | 名称: 30 MM TRISHCL PH 8.0, 0.3 M KCL, 5MM MGCL2, 1MM DTT, 1 MM ATP OR ATP-GAMMA-S pH: 8 詳細: 30 MM TRISHCL PH 8.0, 0.3 M KCL, 5MM MGCL2, 1MM DTT, 1 MM ATP OR ATP-GAMMA-S |
| 試料 | 濃度: 0.07 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: HOLEY CARBON |
| 急速凍結 | 装置: FEI VITROBOT MARK II / 凍結剤: ETHANE / 詳細: LIQUID ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI/PHILIPS CM200FEG / 日付: 2008年4月27日 |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 38000 X / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1500 nm |
| 試料ホルダ | 温度: 82 K |
| 撮影 | 電子線照射量: 15 e/Å2 フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) |
| 画像スキャン | デジタル画像の数: 109 |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: PHASE-FLIPPING | |||||||||||||||
| 3次元再構成 | 手法: IHRSR / 解像度: 17 Å / ピクセルサイズ(公称値): 2.8 Å / ピクセルサイズ(実測値): 2.8 Å 倍率補正: SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2398. (DEPOSITION ID: 11698) 対称性のタイプ: HELICAL | |||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL 詳細: METHOD--RIGID BODY REFINEMENT PROTOCOL--ELECTRON MICROSCPY | |||||||||||||||
| 原子モデル構築 | PDB-ID: 1NY6 Accession code: 1NY6 / Source name: PDB / タイプ: experimental model | |||||||||||||||
| 精密化 | 最高解像度: 17 Å | |||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 17 Å
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




