| 登録情報 | データベース: PDB / ID: 3zyh
|
|---|
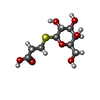
| タイトル | CRYSTAL STRUCTURE OF PA-IL LECTIN COMPLEXED WITH GALBG0 AT 1.5 A RESOLUTION |
|---|
 要素 要素 | PA-I galactophilic lectin |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN/INHIBITOR / SUGAR BINDING PROTEIN-INHIBITOR COMPLEX / ADHESIN / GLYCOSPHINGOLIPID-ANTIGEN / GALACTOSE-SPECIFIC / GALACTOSIDES |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
heterophilic cell-cell adhesion / carbohydrate binding / periplasmic space / cell surface / cytoplasm類似検索 - 分子機能 PA-IL-like / PA-IL-like protein / Galactose-binding lectin / Galactose-binding-like domain superfamily / Jelly Rolls / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 3-(beta-D-galactopyranosylthio)propanoic acid / PA-I galactophilic lectin類似検索 - 構成要素 |
|---|
| 生物種 |   Pseudomonas aeruginosa (緑膿菌) Pseudomonas aeruginosa (緑膿菌)
SYNTHETIC CONSTRUCT (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.501 Å 分子置換 / 解像度: 1.501 Å |
|---|
 データ登録者 データ登録者 | Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. ...Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. / Camara, M. / Stocker, A. / Darbre, T. / Reymond, J.-L. |
|---|
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011
タイトル: A Glycopeptide Dendrimer Inhibitor of the Galactose Specific Lectin Leca and of Pseudomonas Aeruginosa Biofilms
著者: Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. / Camara, M. / Stocker, A. / Darbre, T. / Reymond, J.-L. |
|---|
| 履歴 | | 登録 | 2011年8月23日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2011年9月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年9月28日 | Group: Database references |
|---|
| 改定 1.2 | 2011年10月5日 | Group: Atomic model / Other |
|---|
| 改定 1.3 | 2011年10月26日 | Group: Atomic model |
|---|
| 改定 1.4 | 2011年11月16日 | Group: Database references |
|---|
| 改定 1.5 | 2012年11月30日 | Group: Other |
|---|
| 改定 1.6 | 2015年9月16日 | Group: Source and taxonomy |
|---|
| 改定 1.7 | 2017年8月2日 | Group: Structure summary / カテゴリ: pdbx_molecule_features |
|---|
| 改定 1.8 | 2019年10月2日 | Group: Data collection / Derived calculations ...Data collection / Derived calculations / Experimental preparation / Other
カテゴリ: exptl_crystal_grow / pdbx_database_status ...exptl_crystal_grow / pdbx_database_status / pdbx_struct_assembly / pdbx_struct_assembly_prop
Item: _exptl_crystal_grow.method / _exptl_crystal_grow.temp ..._exptl_crystal_grow.method / _exptl_crystal_grow.temp / _pdbx_database_status.status_code_sf / _pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_assembly_prop.value |
|---|
| 改定 1.9 | 2020年7月29日 | Group: Data collection / Derived calculations
カテゴリ: chem_comp / struct_conn ...chem_comp / struct_conn / struct_site / struct_site_gen
Item: _chem_comp.type / _struct_conn.pdbx_dist_value ..._chem_comp.type / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.10 | 2023年12月20日 | Group: Data collection / Database references ...Data collection / Database references / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.501 Å
分子置換 / 解像度: 1.501 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3zyh.cif.gz
3zyh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3zyh.ent.gz
pdb3zyh.ent.gz PDB形式
PDB形式 3zyh.json.gz
3zyh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3zyh_validation.pdf.gz
3zyh_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3zyh_full_validation.pdf.gz
3zyh_full_validation.pdf.gz 3zyh_validation.xml.gz
3zyh_validation.xml.gz 3zyh_validation.cif.gz
3zyh_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/zy/3zyh
https://data.pdbj.org/pub/pdb/validation_reports/zy/3zyh ftp://data.pdbj.org/pub/pdb/validation_reports/zy/3zyh
ftp://data.pdbj.org/pub/pdb/validation_reports/zy/3zyh リンク
リンク 集合体
集合体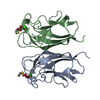
 要素
要素 Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 1
/ ビームライン: X06DA / 波長: 1  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj