+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3zyb | ||||||
|---|---|---|---|---|---|---|---|
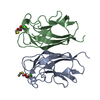
| タイトル | CRYSTAL STRUCTURE OF PA-IL LECTIN COMPLEXED WITH GALAG0 AT 2.3 A RESOLUTION | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | SUGAR BINDING PROTEIN / ADHESIN / GLYCOSPHINGOLIPID-ANTIGEN / GALACTOSE-SPECIFIC / GALACTOSIDES | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heterophilic cell-cell adhesion / carbohydrate binding / periplasmic space / cell surface / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  SYNTHETIC CONSTRUCT (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.29 Å 分子置換 / 解像度: 2.29 Å | ||||||
 データ登録者 データ登録者 | Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. ...Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. / Camara, M. / Stocker, A. / Darbre, T. / Reymond, J.-L. | ||||||
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2011タイトル: A Glycopeptide Dendrimer Inhibitor of the Galactose-Specific Lectin Leca and of Pseudomonas Aeruginosa Biofilms. 著者: Kadam, R.U. / Bergmann, M. / Hurley, M. / Garg, D. / Cacciarini, M. / Swiderska, M.A. / Nativi, C. / Sattler, M. / Smyth, A.R. / Williams, P. / Camara, M. / Stocker, A. / Darbre, T. / Reymond, J.-L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3zyb.cif.gz 3zyb.cif.gz | 217.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3zyb.ent.gz pdb3zyb.ent.gz | 171.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3zyb.json.gz 3zyb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3zyb_validation.pdf.gz 3zyb_validation.pdf.gz | 518.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3zyb_full_validation.pdf.gz 3zyb_full_validation.pdf.gz | 526.4 KB | 表示 | |
| XML形式データ |  3zyb_validation.xml.gz 3zyb_validation.xml.gz | 48.2 KB | 表示 | |
| CIF形式データ |  3zyb_validation.cif.gz 3zyb_validation.cif.gz | 67.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zy/3zyb https://data.pdbj.org/pub/pdb/validation_reports/zy/3zyb ftp://data.pdbj.org/pub/pdb/validation_reports/zy/3zyb ftp://data.pdbj.org/pub/pdb/validation_reports/zy/3zyb | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 7 | 
| ||||||||
| 8 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 / タンパク質・ペプチド / 糖 , 3種, 19分子 ABCDEFGHIJN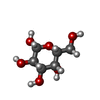

| #1: タンパク質 | 分子量: 12901.333 Da / 分子数: 8 / 由来タイプ: 組換発現 由来: (組換発現)  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 遺伝子: lecA, pa1L, PA2570 / プラスミド: PET25PAIL / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 355.475 Da / 分子数: 3 / 由来タイプ: 合成 / 詳細: GALA-LYS-PRO-LEUNH2 WAS SYNTHESIZED ON SOLID PHASE / 由来: (合成) SYNTHETIC CONSTRUCT (人工物) #4: 糖 | ChemComp-GAL / |
|---|
-非ポリマー , 3種, 959分子 
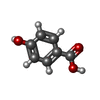



| #3: 化合物 | ChemComp-CA / #5: 化合物 | ChemComp-PHB / #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 48.9 % / 解説: NONE |
|---|---|
| 結晶化 | pH: 3.5 詳細: 0.1 M CITRIC ACID PH 3.5 AND 25% W/V POLYETHYLENE GLYCOL 3350 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06DA / 波長: 1 / ビームライン: X06DA / 波長: 1 |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2010年8月20日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.29→73.07 Å / Num. obs: 45434 / % possible obs: 98.8 % / Observed criterion σ(I): 3 / 冗長度: 3.63 % / Biso Wilson estimate: 30 Å2 / Rmerge(I) obs: 0.1 / Net I/σ(I): 10 |
| 反射 シェル | 解像度: 2.29→2.43 Å / 冗長度: 3.5 % / Rmerge(I) obs: 0.44 / Mean I/σ(I) obs: 2.8 / % possible all: 93.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1L7L 解像度: 2.29→49.261 Å / SU ML: 0.3 / σ(F): 1.99 / 位相誤差: 26.71 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.61 Å / VDWプローブ半径: 0.9 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 37.533 Å2 / ksol: 0.386 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.2 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.29→49.261 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj




