+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3ygs | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | APAF-1 CARD IN COMPLEX WITH PRODOMAIN OF PROCASPASE-9 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | APOPTOSIS / CASPASE ACTIVATION / CASPASE RECRUITMENT / RECOGNITION COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報caspase-9 / response to G1 DNA damage checkpoint signaling / caspase complex / Formation of apoptosome / regulation of apoptotic DNA fragmentation / apoptosome / activation of cysteine-type endopeptidase activity involved in apoptotic process by cytochrome c / leukocyte apoptotic process / glial cell apoptotic process / response to cobalt ion ...caspase-9 / response to G1 DNA damage checkpoint signaling / caspase complex / Formation of apoptosome / regulation of apoptotic DNA fragmentation / apoptosome / activation of cysteine-type endopeptidase activity involved in apoptotic process by cytochrome c / leukocyte apoptotic process / glial cell apoptotic process / response to cobalt ion / cysteine-type endopeptidase activity involved in apoptotic signaling pathway / Caspase activation via Dependence Receptors in the absence of ligand / Activation of caspases through apoptosome-mediated cleavage / Regulation of the apoptosome activity / cysteine-type endopeptidase activity involved in apoptotic process / SMAC (DIABLO) binds to IAPs / SMAC(DIABLO)-mediated dissociation of IAP:caspase complexes / AKT phosphorylates targets in the cytosol / fibroblast apoptotic process / epithelial cell apoptotic process / platelet formation / TP53 Regulates Transcription of Caspase Activators and Caspases / Transcriptional Regulation by E2F6 / Constitutive Signaling by AKT1 E17K in Cancer / cysteine-type endopeptidase activator activity involved in apoptotic process / protein maturation / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / signal transduction in response to DNA damage / forebrain development / cardiac muscle cell apoptotic process / enzyme activator activity / cellular response to transforming growth factor beta stimulus / heat shock protein binding / intrinsic apoptotic signaling pathway / cellular response to dexamethasone stimulus / response to nutrient / kidney development / neural tube closure / response to ischemia / positive regulation of apoptotic signaling pathway / NOD1/2 Signaling Pathway / protein processing / ADP binding / SH3 domain binding / activation of cysteine-type endopeptidase activity involved in apoptotic process / intrinsic apoptotic signaling pathway in response to DNA damage / cellular response to UV / positive regulation of neuron apoptotic process / response to estradiol / nervous system development / peptidase activity / regulation of apoptotic process / secretory granule lumen / neuron apoptotic process / ficolin-1-rich granule lumen / response to lipopolysaccharide / cell differentiation / response to hypoxia / positive regulation of apoptotic process / cysteine-type endopeptidase activity / nucleotide binding / apoptotic process / DNA damage response / Neutrophil degranulation / protein kinase binding / protein-containing complex / mitochondrion / extracellular exosome / extracellular region / ATP binding / identical protein binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  多重同系置換 / 解像度: 2.5 Å 多重同系置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Qin, H. / Srinivasula, S. / Wu, G. / Fernandes-Alnemri, T. / Alnemri, E. / Shi, Y. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 1999 ジャーナル: Nature / 年: 1999タイトル: Structural basis of procaspase-9 recruitment by the apoptotic protease-activating factor 1. 著者: Qin, H. / Srinivasula, S.M. / Wu, G. / Fernandes-Alnemri, T. / Alnemri, E.S. / Shi, Y. | ||||||
| 履歴 |
|
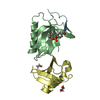
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3ygs.cif.gz 3ygs.cif.gz | 53.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3ygs.ent.gz pdb3ygs.ent.gz | 39.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3ygs.json.gz 3ygs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3ygs_validation.pdf.gz 3ygs_validation.pdf.gz | 369.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3ygs_full_validation.pdf.gz 3ygs_full_validation.pdf.gz | 375.2 KB | 表示 | |
| XML形式データ |  3ygs_validation.xml.gz 3ygs_validation.xml.gz | 6 KB | 表示 | |
| CIF形式データ |  3ygs_validation.cif.gz 3ygs_validation.cif.gz | 9.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yg/3ygs https://data.pdbj.org/pub/pdb/validation_reports/yg/3ygs ftp://data.pdbj.org/pub/pdb/validation_reports/yg/3ygs ftp://data.pdbj.org/pub/pdb/validation_reports/yg/3ygs | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 10925.555 Da / 分子数: 1 / 断片: CASPASE RECRUITMENT DOMAIN (CARD) / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 細胞内の位置: CYTOPLASM / プラスミド: PGEX-2T / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 細胞内の位置: CYTOPLASM / プラスミド: PGEX-2T / 生物種 (発現宿主): Escherichia coli / 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 11312.906 Da / 分子数: 1 / 断片: PRODOMAIN / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 細胞内の位置: CYTOPLASM / プラスミド: PGEX-2T / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 細胞内の位置: CYTOPLASM / プラスミド: PGEX-2T / 生物種 (発現宿主): Escherichia coli / 発現宿主:  参照: UniProt: P55211, 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.8 Å3/Da / 溶媒含有率: 65 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7.5 / 詳細: pH 7.5 | ||||||||||||||||||||
| 結晶 | *PLUS | ||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 |
| 検出器 | 詳細: MIRRORS |
| 放射 | モノクロメーター: NI FILTER / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→99 Å / Num. obs: 134080 / % possible obs: 98.6 % / 冗長度: 11 % / Rmerge(I) obs: 0.056 |
| 反射 シェル | 解像度: 2.5→2.59 Å / Rmerge(I) obs: 0.177 / % possible all: 93.2 |
| 反射 | *PLUS Num. obs: 12452 / Num. measured all: 134080 |
| 反射 シェル | *PLUS % possible obs: 93.2 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多重同系置換 / 解像度: 2.5→10 Å / σ(F): 0 多重同系置換 / 解像度: 2.5→10 Å / σ(F): 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.5→2.61 Å / Total num. of bins used: 10 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.8 / 分類: refinement X-PLOR / バージョン: 3.8 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.5 Å / 最低解像度: 10 Å / σ(F): 0 / % reflection Rfree: 5 % / Rfactor obs: 0.224 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS 最高解像度: 2.5 Å / Rfactor Rfree: 0.46 / % reflection Rfree: 5 % / Rfactor Rwork: 0.35 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj












