| 登録情報 | データベース: PDB / ID: 3vfz
|
|---|
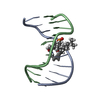
| タイトル | Crystal structure of -35 promoter binding domain of SigD of Mycobacterium tuberculosis |
|---|
 要素 要素 | Probable RNA polymerase sigma-D factor |
|---|
 キーワード キーワード | TRANSCRIPTION / Transcription regulation / Promoter / Anti-sigma factors |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
sigma factor activity / DNA-templated transcription initiation / response to heat / regulation of DNA-templated transcription / DNA binding類似検索 - 分子機能 RNA polymerase sigma factor 70, region 4 type 2 / Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. / RNA polymerase sigma-70 like / RNA polymerase sigma-70 region 2 / RNA polymerase sigma-70 like domain / Sigma-70 region 2 / RNA polymerase sigma factor, region 2 / RNA polymerase sigma factor, region 3/4-like ...RNA polymerase sigma factor 70, region 4 type 2 / Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. / RNA polymerase sigma-70 like / RNA polymerase sigma-70 region 2 / RNA polymerase sigma-70 like domain / Sigma-70 region 2 / RNA polymerase sigma factor, region 2 / RNA polymerase sigma factor, region 3/4-like / Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 ECF RNA polymerase sigma factor SigD / ECF RNA polymerase sigma factor SigD類似検索 - 構成要素 |
|---|
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.901 Å 単波長異常分散 / 解像度: 1.901 Å |
|---|
 データ登録者 データ登録者 | Jaiswal, R.K. / Thakur, K.G. / Gopal, B. |
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2013 ジャーナル: Nucleic Acids Res. / 年: 2013
タイトル: Mycobacterium tuberculosis RsdA provides a conformational rationale for selective regulation of sigma-factor activity by proteolysis
著者: Jaiswal, R.K. / Prabha, T.S. / Manjeera, G. / Gopal, B. |
|---|
| 履歴 | | 登録 | 2012年1月10日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2013年2月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年10月9日 | Group: Database references |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 1.901 Å
単波長異常分散 / 解像度: 1.901 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2013
ジャーナル: Nucleic Acids Res. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3vfz.cif.gz
3vfz.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3vfz.ent.gz
pdb3vfz.ent.gz PDB形式
PDB形式 3vfz.json.gz
3vfz.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3vfz_validation.pdf.gz
3vfz_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3vfz_full_validation.pdf.gz
3vfz_full_validation.pdf.gz 3vfz_validation.xml.gz
3vfz_validation.xml.gz 3vfz_validation.cif.gz
3vfz_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vf/3vfz
https://data.pdbj.org/pub/pdb/validation_reports/vf/3vfz ftp://data.pdbj.org/pub/pdb/validation_reports/vf/3vfz
ftp://data.pdbj.org/pub/pdb/validation_reports/vf/3vfz リンク
リンク 集合体
集合体


 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: BM14 / 波長: 0.978 Å
/ ビームライン: BM14 / 波長: 0.978 Å 解析
解析 単波長異常分散 / 解像度: 1.901→33.883 Å / Occupancy max: 1 / Occupancy min: 1 / SU ML: 0.23 / σ(F): 1 / σ(I): 0 / 位相誤差: 26.61 / 立体化学のターゲット値: ML
単波長異常分散 / 解像度: 1.901→33.883 Å / Occupancy max: 1 / Occupancy min: 1 / SU ML: 0.23 / σ(F): 1 / σ(I): 0 / 位相誤差: 26.61 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

