ソフトウェア 名称 バージョン 分類 データ収集 位相決定 (phenix.refine: 1.7_650)精密化 データ削減 データスケーリング
精密化 解像度 / SU ML / σ(F) / 位相誤差 / 立体化学のターゲット値 Rfactor 反射数 %反射 Rfree 0.2751 520 4.77 % Rwork 0.2194 - - obs 0.2221 10910 89.51 %
溶媒の処理 減衰半径 / VDWプローブ半径 / 溶媒モデル / Bsol 2 / ksol 3 原子変位パラメータ Baniso -1 Baniso -2 Baniso -3 1- -8.7938 Å2 0 Å2 -0 Å2 2- - 34.9925 Å2 -0 Å2 3- - - -26.1987 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 0 2908 88 7 3003
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.006 3328 X-RAY DIFFRACTION f_angle_d1 5175 X-RAY DIFFRACTION f_dihedral_angle_d22.025 1657 X-RAY DIFFRACTION f_chiral_restr0.059 684 X-RAY DIFFRACTION f_plane_restr0.007 141
LS精密化 シェル 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Refine-ID % reflection obs (%)2.6047-2.8661 0.527 138 0.396 2394 X-RAY DIFFRACTION 85 2.8661-3.2793 0.3054 113 0.259 2327 X-RAY DIFFRACTION 82 3.2793-4.1256 0.2371 134 0.2013 2626 X-RAY DIFFRACTION 91 4.1256-20.0336 0.2133 135 0.1699 3043 X-RAY DIFFRACTION 99
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 8) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.0241 0.0231 -0.0319 0.123 0.0523 0.0974 0.2219 -0.1179 -0.1122 -0.0546 0.1691 -0.216 -0.0954 0.0655 0.2387 0.477 0.0767 0.0244 0.8071 0.0908 0.3427 0.4428 -1.3913 22.4553 2 0.3478 -0.1187 -0.4324 0.0423 0.1401 0.551 0.0543 0.1165 -0.0948 -0.2975 0.199 -0.0725 -0.405 0.0181 0.1428 0.4718 0.0345 0.0784 0.6741 -0.0301 0.3627 1.494 0.7691 26.6713 3 0.2746 -0.1983 0.1114 0.89 -0.2949 0.1165 -0.069 -0.3583 -0.131 0.3116 0.1144 0.0115 -0.2457 0.0525 0.5835 0.2651 -0.1303 0.0392 0.4763 -0.0962 0.2456 9.1231 1.8765 -3.9012 4 0.0025 -0.0125 -0.0101 0.2258 0.1681 0.1251 0.1434 -0.0286 -0.0018 0.3396 -0.028 -0.0667 0.151 -0.2359 0.1316 0.3953 -0.0581 -0.0016 0.6876 -0.0633 0.2334 -4.6414 -3.0737 -3.3964 5 0.6503 -0.4128 0.2467 2.0796 0.5598 0.3788 0.1336 -0.0229 0.1209 -0.2582 0.0597 -0.335 -0.1173 0.1272 0.2153 0.4789 -0.0675 0.0872 0.4892 0.2238 0.4561 1.2815 0.523 -68.6889 6 0.0179 -0.1051 -0.0651 0.6423 0.4065 0.2592 0.0939 -0.0943 0.1207 0.1888 0.0154 -0.2554 0.1711 -0.0101 0.195 0.2434 -0.1007 -0.039 0.4231 0.1872 0.3013 2.35 -0.8187 -72.7823 7 0.2394 0.2563 0.1664 0.4223 0.1051 0.1571 -0.096 -0.0375 0.1692 -0.2247 0.2561 0.2113 -0.222 -0.0804 0.228 0.1517 0.0581 -0.0214 0.1444 -0.0307 0.1864 9.597 -3.0693 -41.9137 8 0.0559 -0.0592 -0.0008 0.1162 0.0186 0.059 -0.0623 -0.2075 -0.0047 0.0008 0.1557 -0.1041 0.0115 -0.0746 0.1412 0.0921 0.109 0.2113 0.8398 -0.0742 0.0867 -4.5634 1.211 -42.1991
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details 1 X-RAY DIFFRACTION 1 (chain A and resid 22:30)2 X-RAY DIFFRACTION 2 (chain A and resid 81:89)3 X-RAY DIFFRACTION 3 (chain A and resid 31:47)4 X-RAY DIFFRACTION 4 (chain A and resid 61:80)5 X-RAY DIFFRACTION 5 (chain B and resid 22:30)6 X-RAY DIFFRACTION 6 (chain B and resid 81:89)7 X-RAY DIFFRACTION 7 (chain B and resid 31:47)8 X-RAY DIFFRACTION 8 (chain X and resid 61:80)
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.605 Å
シンクロトロン / 解像度: 2.605 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Chem.Biol. / 年: 2011
ジャーナル: Nat.Chem.Biol. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3skz.cif.gz
3skz.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3skz.ent.gz
pdb3skz.ent.gz PDB形式
PDB形式 3skz.json.gz
3skz.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3skz_validation.pdf.gz
3skz_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3skz_full_validation.pdf.gz
3skz_full_validation.pdf.gz 3skz_validation.xml.gz
3skz_validation.xml.gz 3skz_validation.cif.gz
3skz_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/sk/3skz
https://data.pdbj.org/pub/pdb/validation_reports/sk/3skz ftp://data.pdbj.org/pub/pdb/validation_reports/sk/3skz
ftp://data.pdbj.org/pub/pdb/validation_reports/sk/3skz リンク
リンク 集合体
集合体


 要素
要素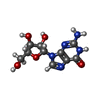








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X29A / 波長: 1.075 Å
/ ビームライン: X29A / 波長: 1.075 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー




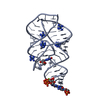
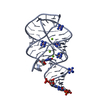
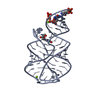







 PDBj
PDBj

































