+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3p4j | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
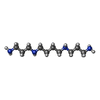
| Title | Ultra-high resolution structure of d(CGCGCG)2 Z-DNA | ||||||||||||||||||
 Components Components | DNA (5'-D(* Keywords KeywordsDNA / Z-DNA | Function / homology | SPERMINE / DNA |  Function and homology information Function and homology informationMethod |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 0.55 Å MOLECULAR REPLACEMENT / Resolution: 0.55 Å  Authors AuthorsBrzezinski, K. / Brzuszkiewicz, A. / Dauter, M. / Kubicki, M. / Jaskolski, M. / Dauter, Z. |  Citation Citation Journal: Nucleic Acids Res. / Year: 2011 Journal: Nucleic Acids Res. / Year: 2011Title: High regularity of Z-DNA revealed by ultra high-resolution crystal structure at 0.55 A. Authors: Brzezinski, K. / Brzuszkiewicz, A. / Dauter, M. / Kubicki, M. / Jaskolski, M. / Dauter, Z. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3p4j.cif.gz 3p4j.cif.gz | 28.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3p4j.ent.gz pdb3p4j.ent.gz | 19.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3p4j.json.gz 3p4j.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/p4/3p4j https://data.pdbj.org/pub/pdb/validation_reports/p4/3p4j ftp://data.pdbj.org/pub/pdb/validation_reports/p4/3p4j ftp://data.pdbj.org/pub/pdb/validation_reports/p4/3p4j | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1ickS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: DNA chain | Mass: 1810.205 Da / Num. of mol.: 2 / Source method: obtained synthetically / Details: Synthetic construct #2: Chemical | ChemComp-SPM / | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.7 Å3/Da / Density % sol: 27.77 % |
|---|---|
| Crystal grow | Temperature: 293 K / pH: 7 Details: DNA water solution mixed 1:1 v/v with 10% MPD, 12 mM spermine tetra-HCl, 40 mM HEPES pH 7.0 and equilibrated against 35% MPD, VAPOR DIFFUSION, HANGING DROP, temperature 293K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 24-ID-C / Wavelength: 0.5904 / Beamline: 24-ID-C / Wavelength: 0.5904 |
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Apr 25, 2010 / Details: FOCUSING MIRRORS |
| Radiation | Monochromator: SI 111 DOUBLE / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.5904 Å / Relative weight: 1 |
| Reflection | Resolution: 0.55→30 Å / Num. obs: 130650 / % possible obs: 84.5 % / Observed criterion σ(I): -3 / Redundancy: 2.5 % / Biso Wilson estimate: 2.45 Å2 / Rmerge(I) obs: 0.057 / Rsym value: 0.057 / Net I/σ(I): 16.7 |
| Reflection shell | Resolution: 0.55→0.57 Å / Redundancy: 1.7 % / Rmerge(I) obs: 0.276 / % possible all: 53 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 1ICK Resolution: 0.55→30 Å / Occupancy max: 1 / Occupancy min: 0.05 / Cross valid method: THROUGHOUT / σ(F): 4 / Stereochemistry target values: NONE Details: USED WEIGHTED FULL MATRIX LEAST SQUARES PROCEDURE AND NON-RESTRAINED ANISOTROPIC REFINEMENT
| ||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 0.55→30 Å
|
 Movie
Movie Controller
Controller










 PDBj
PDBj