+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3mbu | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a bipyridine-modified PNA duplex | |||||||||
 要素 要素 | Bipyridine-PNA | |||||||||
 キーワード キーワード | Peptide Nucleic acid / bipyridine / PNA | |||||||||
| 機能・相同性 | CARBONATE ION / S-1,2-PROPANEDIOL / R-1,2-PROPANEDIOL 機能・相同性情報 機能・相同性情報 | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / AB INITIO PHASING / 解像度: 1.05 Å シンクロトロン / AB INITIO PHASING / 解像度: 1.05 Å | |||||||||
 データ登録者 データ登録者 | Yeh, J.I. / Pohl, E. / Truan, D. / He, W. / Sheldrick, G.M. / Du, S. / Achim, C. | |||||||||
 引用 引用 |  ジャーナル: Chemistry / 年: 2010 ジャーナル: Chemistry / 年: 2010タイトル: The crystal structure of non-modified and bipyridine-modified PNA duplexes. 著者: Yeh, J.I. / Pohl, E. / Truan, D. / He, W. / Sheldrick, G.M. / Du, S. / Achim, C. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3mbu.cif.gz 3mbu.cif.gz | 58.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3mbu.ent.gz pdb3mbu.ent.gz | 48.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3mbu.json.gz 3mbu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mb/3mbu https://data.pdbj.org/pub/pdb/validation_reports/mb/3mbu ftp://data.pdbj.org/pub/pdb/validation_reports/mb/3mbu ftp://data.pdbj.org/pub/pdb/validation_reports/mb/3mbu | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 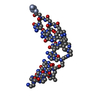
| |||||||||
| 2 | 
| |||||||||
| 3 | 
| |||||||||
| 4 | 
| |||||||||
| 単位格子 |
| |||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質・ペプチド , 1種, 4分子 ABCD
| #1: タンパク質・ペプチド | 分子量: 2620.625 Da / 分子数: 4 / 由来タイプ: 合成 / 詳細: Chemically synthesised |
|---|
-非ポリマー , 6種, 242分子 
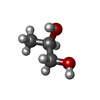


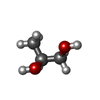






| #2: 化合物 | ChemComp-EDO / | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #3: 化合物 | | #4: 化合物 | #5: 化合物 | #6: 化合物 | #7: 水 | ChemComp-HOH / | |
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 2 X線回折 / 使用した結晶の数: 2 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 48.72 % |
|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法 / pH: 5.5 詳細: 40% propanediol 0.2 M NaCl 0.1 M citrate pH 5.5, VAPOR DIFFUSION, temperature 295K |
-データ収集
| 回折 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| |||||||||||||||
| 検出器 |
| |||||||||||||||
| 放射 |
| |||||||||||||||
| 放射波長 | 波長: 0.8 Å / 相対比: 1 | |||||||||||||||
| 反射 | 解像度: 1.05→50 Å / Num. all: 40632 / Num. obs: 34896 / Observed criterion σ(F): 4 / Observed criterion σ(I): 2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: AB INITIO PHASING / 解像度: 1.05→26.73 Å / 交差検証法: THROUGHOUT / σ(F): 4 / σ(I): -3 / 詳細: conjugate gradient against intensities
| ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.05→26.73 Å
|
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj









