ソフトウェア 名称 バージョン 分類 NB データ削減 Show 3.3.9データスケーリング Show 位相決定 Show refmac_5.5.0102精密化 Show 3.005 データ抽出 Show
精密化 構造決定の手法 / 解像度 / Cor.coef. Fo :Fc / Cor.coef. Fo :Fc free / Occupancy max / Occupancy min / SU B / SU ML / 交差検証法 / σ(F) / ESU R / ESU R Free / 立体化学のターゲット値 詳細 Rfactor 反射数 %反射 Selection details Rfree 0.216 1776 5 % RANDOM Rwork 0.165 - - - obs 0.168 35626 98.96 % -
溶媒の処理 イオンプローブ半径 / 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 Baniso -1 Baniso -2 Baniso -3 1- 8.23 Å2 0 Å2 -10.46 Å2 2- - -6.61 Å2 0 Å2 3- - - -1.62 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 4569 0 76 220 4865
拘束条件 Refine-ID タイプ Dev ideal Dev ideal target 数 X-RAY DIFFRACTION r_bond_refined_d0.013 0.022 4791 X-RAY DIFFRACTION r_angle_refined_deg1.478 2.001 6447 X-RAY DIFFRACTION r_dihedral_angle_1_deg6.756 5 593 X-RAY DIFFRACTION r_dihedral_angle_2_deg42.026 25.369 203 X-RAY DIFFRACTION r_dihedral_angle_3_deg20.057 15 969 X-RAY DIFFRACTION r_dihedral_angle_4_deg18.8 15 25 X-RAY DIFFRACTION r_chiral_restr0.098 0.2 721 X-RAY DIFFRACTION r_gen_planes_refined0.007 0.021 3499 X-RAY DIFFRACTION r_mcbond_it0.473 1.5 2932 X-RAY DIFFRACTION r_mcangle_it0.806 2 4737 X-RAY DIFFRACTION r_scbond_it1.449 3 1859 X-RAY DIFFRACTION r_scangle_it2.229 4.5 1710
LS精密化 シェル 解像度 / Total num. of bins used Rfactor 反射数 %反射 Rfree 0.233 134 - Rwork 0.14 2446 - all - 2580 - obs - - 97.58 %
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 20) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 3.2938 2.0349 0.2808 4.3694 0.1231 2.4598 0.1696 -0.1521 -0.5674 0.4927 -0.1244 -0.0281 0.5687 -0.0705 -0.0452 0.2648 -0.0192 -0.0215 0.034 -0.0036 0.202 -6.5127 -27.9472 2.9764 2 4.9565 -0.6605 -2.842 5.5127 -1.3991 2.5867 -0.0146 0.8597 0.4358 -1.0353 0.2909 0.072 0.0719 -0.3925 -0.2763 0.3702 -0.1548 -0.1125 0.2926 -0.0045 0.2115 -6.1763 -15.5809 -8.533 3 3.2817 1.6967 0.5639 4.7318 0.2937 2.3793 0.1034 0.02 -0.1919 0.3599 -0.0783 0.2082 0.2047 -0.0348 -0.0252 0.1756 -0.0281 -0.0052 0.0005 -0.0182 0.0838 -6.0512 -21.9703 4.0002 4 1.0485 0.6949 -1.1446 11.426 1.6881 1.9343 -0.2131 0.4195 -0.5338 -0.4447 0.1203 -1.0655 0.0259 0.0667 0.0929 0.2712 -0.0708 0.1073 0.5936 -0.264 0.5501 18.4138 -15.8547 -6.7205 5 3.5151 2.0466 0.057 5.085 1.4073 3.812 0.0114 0.0028 -0.163 0.1356 0.0491 -0.377 -0.3498 0.2315 -0.0605 0.1557 -0.0861 -0.0755 0.1103 0.0386 0.1253 11.3036 -4.6444 6.3161 6 4.2382 -1.3167 -2.771 4.6661 4.6244 5.4798 -0.1749 -0.2995 -0.3169 1.0353 0.1522 -0.3156 0.7391 0.4663 0.0227 0.3697 -0.1256 -0.1271 0.1587 0.0442 0.3095 12.7114 -6.5383 12.2453 7 1.1734 1.7999 0.7437 2.5672 1.3023 4.3152 0.1183 -0.0506 0.1081 0.2238 -0.0849 -0.0767 -0.1439 0.1545 -0.0334 0.1861 -0.018 -0.0795 0.0544 0.0257 0.2135 9.1792 1.3524 8.5916 8 15.6378 5.044 12.4306 1.1335 3.2406 10.7606 -0.0601 0.0855 1.4147 0.1252 -0.0056 0.5657 -0.6061 -0.2037 0.0657 0.5064 -0.0208 0.0418 0.1667 0.2092 0.7409 6.6315 11.5889 1.4784 9 2.2395 2.0105 -0.4381 5.8171 -0.6167 1.3682 -0.1377 0.4659 -0.0108 -0.2273 0.228 -0.2302 -0.3778 0.378 -0.0902 0.2065 -0.1386 0.03 0.2232 -0.0182 0.0919 10.7924 -0.7553 -0.552 10 6.7309 5.22 -5.4058 22.2332 -3.8585 2.0849 -0.4678 1.0565 -0.1064 -0.8075 0.5149 -0.0448 0.0568 -0.4143 -0.0471 0.5451 -0.5359 0.1155 0.7224 -0.1552 0.1686 14.2575 -8.0052 -8.7851 11 4.0851 0.0728 2.1571 6.7179 0.4933 2.2704 -0.2461 0.0793 0.5938 -0.1929 -0.0935 0.6276 -0.6763 -0.1545 0.3397 0.3767 0.0531 -0.0645 0.0924 -0.0133 0.2138 6.8308 -29.0924 28.1938 12 1.4131 -0.9016 -1.6004 4.3514 1.5537 4.2958 -0.0164 -0.3944 -0.3023 0.71 0.0495 -0.6475 0.2951 0.7372 -0.0331 0.2804 0.0171 -0.1327 0.1674 0.0714 0.2547 17.3993 -43.735 32.0274 13 6.0893 -1.5286 -2.0657 1.3912 2.4661 3.3919 -0.3643 -1.0664 -0.274 0.6735 0.0984 0.2886 0.7051 0.0693 0.2659 0.6119 -0.0078 0.2139 0.1674 0.0179 0.2246 4.3353 -41.4928 35.7714 14 0.3533 -1.764 -0.1591 5.8134 1.8108 5.5734 -0.0463 0.1876 -0.0074 -0.0961 -0.2798 0.7775 -0.4423 -0.5757 0.3261 0.2063 0.0343 -0.0633 0.0851 0.078 0.4175 4.9696 -38.7679 23.1326 15 2.3613 -0.795 0.0353 3.043 2.2131 2.85 0.2132 0.2338 0.4077 -0.7537 0.1444 -0.9513 -0.5535 0.4418 -0.3577 0.3863 -0.0715 0.1687 0.239 -0.0044 0.3927 24.6801 -40.8815 12.4886 16 4.739 -0.9229 0.5395 6.2191 1.867 4.0859 0.0143 0.0094 -0.2828 -0.3446 0.0899 0.4555 0.0374 -0.3026 -0.1042 0.2143 0.0128 -0.0291 0.092 0.0155 0.0653 11.656 -55.1632 12.8174 17 8.5283 -0.9622 4.4511 1.0286 2.077 6.4076 0.3495 1.058 0.323 -0.4297 -0.403 0.2254 -0.2033 -0.0536 0.0535 0.3762 0.0848 -0.1781 0.1284 0.0161 0.4052 8.1478 -53.9571 7.8584 18 1.7491 -0.7819 2.007 6.2192 0.3781 1.9247 -0.1304 -0.0059 -0.2766 -0.2867 0.1957 0.3685 -0.0455 0.0805 -0.0653 0.2367 0.0455 0.0337 0.073 -0.0301 0.1758 10.7992 -61.7373 10.5322 19 1.3679 -0.9256 0.3349 6.6823 1.851 2.6824 0.2078 0.1281 -0.3897 0.5658 0.118 -0.1488 0.7421 0.1914 -0.3258 0.2777 0.0274 -0.0899 0.1411 -0.0516 0.1877 17.8607 -66.4918 14.3259 20 2.9786 -0.7597 -0.8955 1.3612 1.2988 7.6716 -0.0286 -0.2302 -0.057 -0.2332 0.4369 -0.6743 0.5032 1.2476 -0.4083 0.2188 -0.0229 0.1176 0.2662 -0.1546 0.3952 28.0667 -52.9199 14.6814
精密化 TLSグループ 大きな表を表示 (5 x 20) 大きな表を隠す ID Refine-ID Refine TLS-ID Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 A36 - 68 2 X-RAY DIFFRACTION 2 A69 - 98 3 X-RAY DIFFRACTION 3 A99 - 173 4 X-RAY DIFFRACTION 4 A174 - 191 5 X-RAY DIFFRACTION 5 A192 - 219 6 X-RAY DIFFRACTION 6 A220 - 239 7 X-RAY DIFFRACTION 7 A240 - 267 8 X-RAY DIFFRACTION 8 A268 - 283 9 X-RAY DIFFRACTION 9 A284 - 310 10 X-RAY DIFFRACTION 10 A311 - 323 11 X-RAY DIFFRACTION 11 B36 - 61 12 X-RAY DIFFRACTION 12 B62 - 101 13 X-RAY DIFFRACTION 13 B102 - 122 14 X-RAY DIFFRACTION 14 B123 - 161 15 X-RAY DIFFRACTION 15 B162 - 196 16 X-RAY DIFFRACTION 16 B197 - 219 17 X-RAY DIFFRACTION 17 B220 - 240 18 X-RAY DIFFRACTION 18 B241 - 266 19 X-RAY DIFFRACTION 19 B267 - 305 20 X-RAY DIFFRACTION 20 B306 - 327
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2010
ジャーナル: J.Biol.Chem. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3li2.cif.gz
3li2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3li2.ent.gz
pdb3li2.ent.gz PDB形式
PDB形式 3li2.json.gz
3li2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3li2_validation.pdf.gz
3li2_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3li2_full_validation.pdf.gz
3li2_full_validation.pdf.gz 3li2_validation.xml.gz
3li2_validation.xml.gz 3li2_validation.cif.gz
3li2_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/li/3li2
https://data.pdbj.org/pub/pdb/validation_reports/li/3li2 ftp://data.pdbj.org/pub/pdb/validation_reports/li/3li2
ftp://data.pdbj.org/pub/pdb/validation_reports/li/3li2 リンク
リンク 集合体
集合体
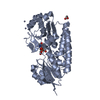

 要素
要素


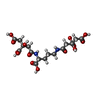







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CLSI
CLSI  / ビームライン: 08ID-1 / 波長: 0.97934 Å
/ ビームライン: 08ID-1 / 波長: 0.97934 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 2.2→33.15 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.93 / Occupancy max: 1 / Occupancy min: 0.5 / SU B: 11.226 / SU ML: 0.136 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.046 / ESU R Free: 0.039 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
分子置換 / 解像度: 2.2→33.15 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.93 / Occupancy max: 1 / Occupancy min: 0.5 / SU B: 11.226 / SU ML: 0.136 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.046 / ESU R Free: 0.039 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj








