| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| HKL-2000 | | データ収集 | | PHASER | | 位相決定 | | PHENIX | (phenix.refine: 1.5_2)| 精密化 | | DENZO | | データ削減 | | SCALEPACK | | データスケーリング | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: PDB ENTRIES; 2JTM, 1AZP
解像度: 1.602→16.732 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.846 / SU ML: 0.14 / σ(F): 0.02 / 位相誤差: 21.95 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.217 | 765 | 5.09 % |
|---|
| Rwork | 0.1759 | 14277 | - |
|---|
| obs | 0.178 | 15042 | 90.61 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 58.385 Å2 / ksol: 0.434 e/Å3 |
|---|
| 原子変位パラメータ | Biso max: 123.87 Å2 / Biso mean: 41.316 Å2 / Biso min: 17.81 Å2
| Baniso -1 | Baniso -2 | Baniso -3 |
|---|
| 1- | -5.455 Å2 | 0 Å2 | -0 Å2 |
|---|
| 2- | - | 7.355 Å2 | 0 Å2 |
|---|
| 3- | - | - | -1.9 Å2 |
|---|
|
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 1.602→16.732 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 445 | 322 | 0 | 135 | 902 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.01 | 818 | | X-RAY DIFFRACTION | f_angle_d| 1.613 | 1168 | | X-RAY DIFFRACTION | f_chiral_restr| 0.073 | 126 | | X-RAY DIFFRACTION | f_plane_restr| 0.007 | 93 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 24.35 | 334 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 1.6018-1.7253 | 0.2398 | 114 | 0.2024 | 2421 | X-RAY DIFFRACTION | 79 | | 1.7253-1.8987 | 0.2234 | 142 | 0.1896 | 2777 | X-RAY DIFFRACTION | 88 | | 1.8987-2.173 | 0.231 | 158 | 0.1819 | 3027 | X-RAY DIFFRACTION | 97 | | 2.173-2.7358 | 0.2281 | 180 | 0.1832 | 3030 | X-RAY DIFFRACTION | 97 | | 2.7358-16.7334 | 0.2044 | 171 | 0.1656 | 3022 | X-RAY DIFFRACTION | 92 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 5.6056 | 0.4209 | 1.1041 | 5.5998 | 2.4116 | 1.2261 | -0.166 | -1.0626 | 0.279 | 0.2359 | 0.3047 | -0.0982 | -0.2086 | 0.0172 | -0.131 | 0.2958 | -0.0132 | 0.0037 | 0.4204 | -0.0526 | 0.2394 | 7.6747 | 18.7517 | 22.4542 | | 2 | 7.5969 | 5.7163 | 5.494 | 6.1871 | 7.4633 | 9.8402 | -0.4417 | -0.8954 | 0.9169 | -0.1268 | 0.3072 | 0.5279 | -1.3667 | 0.5957 | 0.0396 | 0.3557 | -0.0438 | -0.0117 | 0.2237 | -0.1094 | 0.2879 | 5.8959 | 24.9436 | 20.4358 | | 3 | 1.8566 | 0.0237 | 0.4907 | 0.3376 | 0.1223 | 1.0666 | -0.0489 | -0.187 | -0.2021 | 0.2109 | 0.0474 | -0.0668 | 0.3032 | 0.1608 | -0.02 | 0.2755 | 0.0686 | -0.0021 | 0.2105 | 0.0052 | 0.2371 | 9.9156 | 10.8747 | 13.6388 | | 4 | 2.0066 | -0.7117 | 0.668 | 2.3187 | 0.7515 | 2.3982 | -0.2075 | -0.007 | 0.0657 | -0.1704 | -0.0023 | -0.0973 | -0.3041 | -0.1544 | 0.1854 | 0.2663 | 0.0789 | -0.0152 | 0.2599 | -0.0155 | 0.2022 | 4.4209 | 20.2541 | 14.0456 | | 5 | 4.6489 | -1.2697 | 0.0842 | 1.7777 | -1.4479 | 4.4328 | -0.1452 | -0.8916 | -0.0284 | 0.0576 | 0.0824 | -0.3779 | 0.5768 | 0.8834 | 0.2158 | 0.3957 | 0.1639 | -0.0486 | 0.5246 | 0.0681 | 0.3032 | 12.9773 | 9.2357 | 22.2502 | | 6 | 3.4306 | -1.2323 | -0.4271 | 4.9795 | 1.6502 | 0.5894 | -0.1547 | 0.4093 | -0.4591 | -0.4867 | 0.0885 | -0.3415 | 0.402 | 0.275 | 0.1003 | 0.323 | 0.0664 | 0.0476 | 0.3224 | -0.0299 | 0.2747 | 15.5291 | 7.3667 | 2.8963 | | 7 | 4.7953 | -1.7537 | -0.0756 | 1.0286 | 0.8998 | 1.9244 | -0.0371 | 0.3506 | -0.3895 | 0.0225 | -0.139 | 0.3553 | 0.0275 | -0.1911 | 0.1431 | 0.2815 | 0.0178 | -0.034 | 0.2407 | -0.0192 | 0.2425 | 0.7179 | 12.202 | 6.1358 | | 8 | 2.9373 | 0.9947 | 2.2566 | 2.4387 | 3.0505 | 4.1938 | -0.0138 | 1.2401 | -0.0484 | -0.4963 | 0.118 | -0.3576 | -0.7171 | 0.8446 | -0.1111 | 0.3699 | -0.0043 | -0.063 | 0.4792 | -0.0284 | 0.2732 | 5.5326 | 15.2645 | -0.9116 | | 9 | 7.0734 | -3.1032 | -3.7646 | 2.2001 | 3.1881 | 5.2609 | -0.2178 | -0.4714 | 0.7545 | -0.0759 | 0.4185 | -0.3416 | -0.026 | 0.9249 | -0.2446 | 0.2568 | 0.0387 | 0.0365 | 0.3761 | -0.026 | 0.2816 | 18.5023 | 15.0393 | 8.7419 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details | Auth asym-ID | Auth seq-ID |
|---|
| 1 | X-RAY DIFFRACTION | 1 | (chain A and resid 5:12)A| 5 - 12 | | 2 | X-RAY DIFFRACTION | 2 | (chain A and resid 13:21)A| 13 - 21 | | 3 | X-RAY DIFFRACTION | 3 | (chain A and resid 22:44)A| 22 - 44 | | 4 | X-RAY DIFFRACTION | 4 | (chain A and resid 45:54)A| 45 - 54 | | 5 | X-RAY DIFFRACTION | 5 | (chain A and resid 55:60)A| 55 - 60 | | 6 | X-RAY DIFFRACTION | 6 | (chain B and resid 101:104)B| 101 - 104 | | 7 | X-RAY DIFFRACTION | 7 | (chain B and resid 105:108)B| 105 - 108 | | 8 | X-RAY DIFFRACTION | 8 | (chain C and resid 109:112)C| 109 - 112 | | 9 | X-RAY DIFFRACTION | 9 | (chain C and resid 113:116)C| 113 - 116 | | | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
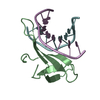
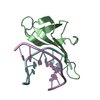
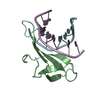
 Sulfolobus solfataricus (古細菌)
Sulfolobus solfataricus (古細菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.602 Å
分子置換 / 解像度: 1.602 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Protein Sci. / 年: 2010
ジャーナル: Protein Sci. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3kxt.cif.gz
3kxt.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3kxt.ent.gz
pdb3kxt.ent.gz PDB形式
PDB形式 3kxt.json.gz
3kxt.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3kxt_validation.pdf.gz
3kxt_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3kxt_full_validation.pdf.gz
3kxt_full_validation.pdf.gz 3kxt_validation.xml.gz
3kxt_validation.xml.gz 3kxt_validation.cif.gz
3kxt_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kx/3kxt
https://data.pdbj.org/pub/pdb/validation_reports/kx/3kxt ftp://data.pdbj.org/pub/pdb/validation_reports/kx/3kxt
ftp://data.pdbj.org/pub/pdb/validation_reports/kx/3kxt リンク
リンク 集合体
集合体
 要素
要素
 Sulfolobus solfataricus (古細菌) / 遺伝子: SSO6901 / プラスミド: pET30a / 発現宿主:
Sulfolobus solfataricus (古細菌) / 遺伝子: SSO6901 / プラスミド: pET30a / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 0.9796 Å
/ ビームライン: BL17U / 波長: 0.9796 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj
