+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3crf | ||||||
|---|---|---|---|---|---|---|---|
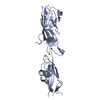
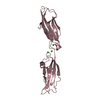
| タイトル | Electron Microscopy model of the Saf Pilus- Type B | ||||||
 要素 要素 | (Outer membrane protein) x 3 | ||||||
 キーワード キーワード | MEMBRANE PROTEIN / SafA protein polymer / Type B Saf Pilus / Fibril Protein | ||||||
| 機能・相同性 | Saf-pilin pilus formation protein / Saf-pilin pilus formation protein / Dr-adhesin superfamily / Adhesion domain superfamily / Prokaryotic membrane lipoprotein lipid attachment site profile. / Outer membrane protein 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌) | ||||||
| 手法 | 電子顕微鏡法 / らせん対称体再構成法 / ネガティブ染色法 / 解像度: 17 Å | ||||||
 データ登録者 データ登録者 | Salih, O. / Remaut, H. / Waksman, G. / Orlova, E.V. | ||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2008 ジャーナル: J Mol Biol / 年: 2008タイトル: Structural analysis of the Saf pilus by electron microscopy and image processing. 著者: Osman Salih / Han Remaut / Gabriel Waksman / Elena V Orlova /  要旨: Bacterial pili are important virulence factors involved in host cell attachment and/or biofilm formation, key steps in establishing and maintaining successful infection. Here we studied Salmonella ...Bacterial pili are important virulence factors involved in host cell attachment and/or biofilm formation, key steps in establishing and maintaining successful infection. Here we studied Salmonella atypical fimbriae (or Saf pili), formed by the conserved chaperone/usher pathway. In contrast to the well-established quaternary structure of typical/FGS-chaperone assembled, rod-shaped, chaperone/usher pili, little is known about the supramolecular organisation in atypical/FGL-chaperone assembled fimbriae. In our study, we have used negative stain electron microscopy and single-particle image analysis to determine the three-dimensional structure of the Salmonella typhimurium Saf pilus. Our results show atypical/FGL-chaperone assembled fimbriae are composed of highly flexible linear multi-subunit fibres that are formed by globular subunits connected to each other by short links giving a "beads on a string"-like appearance. Quantitative fitting of the atomic structure of the SafA pilus subunit into the electron density maps, in combination with linker modelling and energy minimisation, has enabled analysis of subunit arrangement and intersubunit interactions in the Saf pilus. Short intersubunit linker regions provide the molecular basis for flexibility of the Saf pilus by acting as molecular hinges allowing a large range of movement between consecutive subunits in the fibre. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3crf.cif.gz 3crf.cif.gz | 57.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3crf.ent.gz pdb3crf.ent.gz | 40.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3crf.json.gz 3crf.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3crf_validation.pdf.gz 3crf_validation.pdf.gz | 609.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3crf_full_validation.pdf.gz 3crf_full_validation.pdf.gz | 614.2 KB | 表示 | |
| XML形式データ |  3crf_validation.xml.gz 3crf_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  3crf_validation.cif.gz 3crf_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cr/3crf https://data.pdbj.org/pub/pdb/validation_reports/cr/3crf ftp://data.pdbj.org/pub/pdb/validation_reports/cr/3crf ftp://data.pdbj.org/pub/pdb/validation_reports/cr/3crf | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12992.533 Da / 分子数: 1 断片: core pilin domain, N-terminal deleted, UNP residues 48-170 由来タイプ: 組換発現 由来: (組換発現)  Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌)株: LT2 / 遺伝子: safA / プラスミド: PTRC99A / 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 15270.004 Da / 分子数: 1 / 断片: core pilin domain, UNP residues 27-170 / 由来タイプ: 組換発現 由来: (組換発現)  Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌)株: LT2 / 遺伝子: safA / プラスミド: PTRC99A / 発現宿主:  |
| #3: タンパク質・ペプチド | 分子量: 2070.237 Da / 分子数: 1 / 断片: N-TERMINAL EXTENSION, UNP residues 27-45 / 由来タイプ: 合成 / 詳細: SafANte-N-terminal extension / 参照: UniProt: Q8ZRK4 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: らせん対称体再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Wild-type SafA pili / タイプ: COMPLEX 詳細: Stained with 2% ammonium molybdate. Type B Saf pilus. Fibre. |
|---|---|
| 緩衝液 | 名称: 10 mM TRIS-HCl (pH 8) and 10 mM NaCl / pH: 8 / 詳細: 10 mM TRIS-HCl (pH 8) and 10 mM NaCl |
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色: YES / 凍結: NO |
| 染色 | タイプ: NEGATIVE / 染色剤: Ammonium Molybdate |
| 試料支持 | 詳細: Continuous-carbon film |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI TECNAI 10 / 日付: 2004年4月23日 |
|---|---|
| 電子銃 | 電子線源: TUNGSTEN HAIRPIN / 加速電圧: 100 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 44000 X / 最大 デフォーカス(公称値): 900 nm / 最小 デフォーカス(公称値): 400 nm / Cs: 2.4 mm |
| 撮影 | 電子線照射量: 25 e/Å2 / フィルム・検出器のモデル: KODAK SO-163 FILM |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||
| 3次元再構成 | 手法: Cross-common lines / 解像度: 17 Å / 粒子像の数: 8500 / ピクセルサイズ(実測値): 1.591 Å / 対称性のタイプ: POINT | ||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: RECIPROCAL / Target criteria: Correlation coefficient / 詳細: REFINEMENT PROTOCOL--Rigid body | ||||||||||||
| 原子モデル構築 | PDB-ID: 2CO4 Accession code: 2CO4 / Source name: PDB / タイプ: experimental model | ||||||||||||
| 精密化ステップ | サイクル: LAST
|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera
















 PDBj
PDBj




