+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3bpr | ||||||
|---|---|---|---|---|---|---|---|
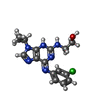
| タイトル | Crystal structure of catalytic domain of the proto-oncogene tyrosine-protein kinase MER in complex with inhibitor C52 | ||||||
 要素 要素 | Proto-oncogene tyrosine-protein kinase MER | ||||||
 キーワード キーワード | TRANSFERASE / ATP-BINDING / DISEASE MUTATION / GLYCOPROTEIN / KINASE / NUCLEOTIDE-BINDING / PHOSPHORYLATION / PROTO-ONCOGENE / RECEPTOR / RETINITIS PIGMENTOSA / SENSORY TRANSDUCTION / TYROSINE-PROTEIN KINASE / VISION / STRUCTURAL GENOMICS / STRUCTURAL GENOMICS CONSORTIUM / SGC / Immunoglobulin domain / Membrane / Phosphoprotein / Polymorphism / Transmembrane | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of leukocyte apoptotic process / negative regulation of lymphocyte activation / neutrophil clearance / natural killer cell differentiation / secretion by cell / negative regulation of cytokine production / vagina development / photoreceptor outer segment / phagocytosis / transmembrane receptor protein tyrosine kinase activity ...negative regulation of leukocyte apoptotic process / negative regulation of lymphocyte activation / neutrophil clearance / natural killer cell differentiation / secretion by cell / negative regulation of cytokine production / vagina development / photoreceptor outer segment / phagocytosis / transmembrane receptor protein tyrosine kinase activity / substrate adhesion-dependent cell spreading / positive regulation of phagocytosis / cell surface receptor protein tyrosine kinase signaling pathway / Cell surface interactions at the vascular wall / establishment of localization in cell / receptor protein-tyrosine kinase / platelet activation / cell migration / cell-cell signaling / nervous system development / retina development in camera-type eye / spermatogenesis / cell surface receptor signaling pathway / protein phosphorylation / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / receptor complex / extracellular space / ATP binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Walker, J.R. / Huang, X. / Finerty Jr, P.J. / Weigelt, J. / Arrowsmith, C.H. / Edwards, A.M. / Bochkarev, A. / Dhe-Paganon, S. / Structural Genomics Consortium (SGC) | ||||||
 引用 引用 |  ジャーナル: J.Struct.Biol. / 年: 2009 ジャーナル: J.Struct.Biol. / 年: 2009タイトル: Structural insights into the inhibited states of the Mer receptor tyrosine kinase. 著者: Huang, X. / Finerty, P. / Walker, J.R. / Butler-Cole, C. / Vedadi, M. / Schapira, M. / Parker, S.A. / Turk, B.E. / Thompson, D.A. / Dhe-Paganon, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3bpr.cif.gz 3bpr.cif.gz | 426.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3bpr.ent.gz pdb3bpr.ent.gz | 352.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3bpr.json.gz 3bpr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bp/3bpr https://data.pdbj.org/pub/pdb/validation_reports/bp/3bpr ftp://data.pdbj.org/pub/pdb/validation_reports/bp/3bpr ftp://data.pdbj.org/pub/pdb/validation_reports/bp/3bpr | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / End auth comp-ID: ASP / End label comp-ID: ASP / Refine code: 1
|
- 要素
要素
| #1: タンパク質 | 分子量: 35889.434 Da / 分子数: 4 / 断片: Catalytic domain: Residues 574-864 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MERTK, MER / プラスミド: pET28a-LIC / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MERTK, MER / プラスミド: pET28a-LIC / 生物種 (発現宿主): Escherichia coli / 発現宿主:  #2: 化合物 | ChemComp-OLP / #3: 化合物 | ChemComp-NA / | #4: 化合物 | #5: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.69 Å3/Da / 溶媒含有率: 54.32 % |
|---|---|
| 結晶化 | 温度: 287 K / pH: 8.5 詳細: Compound C52 (50 mM in DMSO) was added to 8 mg/ml MER protein to the final concentration of 2.5 mM. The mixture was rocked at 277 K overnight and further concentrated to about 35 mg/ml. ...詳細: Compound C52 (50 mM in DMSO) was added to 8 mg/ml MER protein to the final concentration of 2.5 mM. The mixture was rocked at 277 K overnight and further concentrated to about 35 mg/ml. Crystals were grown by mixing 2 microliters of MER inhibitor solution and 2 microliters of reservoir solution (100 mM Tris-HCl pH 8.5, 3.64 M NaCl) at 287 K using the hanging-drop vapor-diffusion method. Crystals were cryo-protected with a solution composed of glycerol, ethylene glycol, glucose, and fructose., VAPOR DIFFUSION, HANGING DROP, pH 8.50 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 17-ID / 波長: 1 / ビームライン: 17-ID / 波長: 1 |
| 検出器 | タイプ: ADSC QUANTUM 210 / 検出器: CCD / 日付: 2007年3月8日 / 詳細: MIRRORS |
| 放射 | モノクロメーター: CRYO-COOLED SI(111) DOUBLE- CRYSTAL プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→50 Å / Num. obs: 37537 / % possible obs: 99 % / Observed criterion σ(I): -3 / 冗長度: 3.3 % / Rsym value: 0.15 / Net I/σ(I): 9.1 |
| 反射 シェル | 解像度: 2.8→2.9 Å / 冗長度: 2.9 % / Mean I/σ(I) obs: 2.3 / Rsym value: 0.53 / % possible all: 94 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 2P0C 解像度: 2.8→47.3 Å / Cor.coef. Fo:Fc: 0.918 / Cor.coef. Fo:Fc free: 0.894 / SU B: 39.32 / SU ML: 0.385 / 交差検証法: THROUGHOUT / ESU R: 1.719 / ESU R Free: 0.42 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. ATOM RECORD CONTAINS SUM OF TLS AND RESIDUAL B FACTORS, ANISOU RECORD CONTAINS SUM OF TLS AND RESIDUAL U FACTORS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 42.487 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→47.3 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Ens-ID: 1 / 数: 1932 / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.872 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj