| 登録情報 | データベース: PDB / ID: 2vyt
|
|---|
| タイトル | The MBT repeats of human SCML2 bind to peptides containing mono methylated lysine. |
|---|
 要素 要素 | SEX COMB ON MIDLEG-LIKE PROTEIN 2 |
|---|
 キーワード キーワード | TRANSCRIPTION / MONO METHYLATED PEPTIDES / MBT REPEATS / PHOSPHOPROTEIN / ALTERNATIVE SPLICING / TRANSCRIPTION REGULATION / NUCLEUS / REPRESSOR / CHROMATIN / HUMAN SCML2 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
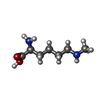
PcG protein complex / anatomical structure morphogenesis / histone binding / negative regulation of DNA-templated transcription / chromatin binding / nucleus類似検索 - 分子機能 Polycomb group protein, RNA binding region / RNA binding Region / : / SLED domain / SLED domain superfamily / SLED domain / Mbt repeat / MBT repeat profile. / Present in Drosophila Scm, l(3)mbt, and vertebrate SCML2 / : ...Polycomb group protein, RNA binding region / RNA binding Region / : / SLED domain / SLED domain superfamily / SLED domain / Mbt repeat / MBT repeat profile. / Present in Drosophila Scm, l(3)mbt, and vertebrate SCML2 / : / mbt repeat / SAM domain (Sterile alpha motif) / SH3 type barrels. - #140 / Sterile alpha motif. / Sterile alpha motif domain / Sterile alpha motif/pointed domain superfamily / SH3 type barrels. / Roll / Mainly Beta類似検索 - ドメイン・相同性 N-METHYL-LYSINE / TRIETHYLENE GLYCOL / Sex comb on midleg-like protein 2類似検索 - 構成要素 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Santiveri, C.M. / Lechtenberg, B.C. / Allen, M.D. / Sathyamurthy, A. / Jaulent, A.M. / Freund, S.M.V. / Bycroft, M. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2008 ジャーナル: J.Mol.Biol. / 年: 2008
タイトル: The Malignant Brain Tumor Repeats of Human Scml2 Bind to Peptides Containing Monomethylated Lysine.
著者: Santiveri, C.M. / Lechtenberg, B.C. / Allen, M.D. / Sathyamurthy, A. / Jaulent, A.M. / Freund, S.M.V. / Bycroft, M. |
|---|
| 履歴 | | 登録 | 2008年7月28日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2008年8月5日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年5月7日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2018年1月24日 | Group: Source and taxonomy / カテゴリ: entity_src_gen
Item: _entity_src_gen.pdbx_host_org_ncbi_taxonomy_id / _entity_src_gen.pdbx_host_org_scientific_name ..._entity_src_gen.pdbx_host_org_ncbi_taxonomy_id / _entity_src_gen.pdbx_host_org_scientific_name / _entity_src_gen.pdbx_host_org_strain / _entity_src_gen.pdbx_host_org_variant |
|---|
| 改定 1.4 | 2023年12月13日 | Group: Data collection / Database references ...Data collection / Database references / Other / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2008
ジャーナル: J.Mol.Biol. / 年: 2008 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2vyt.cif.gz
2vyt.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2vyt.ent.gz
pdb2vyt.ent.gz PDB形式
PDB形式 2vyt.json.gz
2vyt.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2vyt_validation.pdf.gz
2vyt_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2vyt_full_validation.pdf.gz
2vyt_full_validation.pdf.gz 2vyt_validation.xml.gz
2vyt_validation.xml.gz 2vyt_validation.cif.gz
2vyt_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vy/2vyt
https://data.pdbj.org/pub/pdb/validation_reports/vy/2vyt ftp://data.pdbj.org/pub/pdb/validation_reports/vy/2vyt
ftp://data.pdbj.org/pub/pdb/validation_reports/vy/2vyt リンク
リンク 集合体
集合体

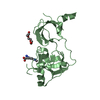
 要素
要素 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-2 / 波長: 0.954
/ ビームライン: ID14-2 / 波長: 0.954  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj