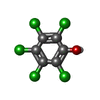
登録情報 データベース : PDB / ID : 2vpyタイトル Polysulfide reductase with bound quinone inhibitor, pentachlorophenol (PCP) HYPOTHETICAL MEMBRANE SPANNING PROTEIN NRFC PROTEIN THIOSULFATE REDUCTASE キーワード / / / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 THERMUS THERMOPHILUS (バクテリア)手法 / / / 解像度 : 2.5 Å データ登録者 Jormakka, M. / Yokoyama, K. / Yano, T. / Tamakoshi, M. / Akimoto, S. / Shimamura, T. / Curmi, P. / Iwata, S. ジャーナル : Nat.Struct.Mol.Biol. / 年 : 2008タイトル : Molecular Mechanism of Energy Conservation in Polysulfide Respiration.著者 : Jormakka, M. / Yokoyama, K. / Yano, T. / Tamakoshi, M. / Akimoto, S. / Shimamura, T. / Curmi, P. / Iwata, S. 履歴 登録 2008年3月9日 登録サイト / 処理サイト 改定 1.0 2008年6月10日 Provider / タイプ 改定 1.1 2011年5月8日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2024年5月8日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Other カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_struct_conn_angle / struct_conn Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id
すべて表示 表示を減らす Remark 700 SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AG" IN EACH CHAIN ON SHEET RECORDS BELOW ... SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AG" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 6-STRANDED BARREL THIS IS REPRESENTED BY A 7-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 THERMUS THERMOPHILUS (バクテリア)
THERMUS THERMOPHILUS (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2.5 Å
多波長異常分散 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2008
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2008 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2vpy.cif.gz
2vpy.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2vpy.ent.gz
pdb2vpy.ent.gz PDB形式
PDB形式 2vpy.json.gz
2vpy.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2vpy_validation.pdf.gz
2vpy_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2vpy_full_validation.pdf.gz
2vpy_full_validation.pdf.gz 2vpy_validation.xml.gz
2vpy_validation.xml.gz 2vpy_validation.cif.gz
2vpy_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vp/2vpy
https://data.pdbj.org/pub/pdb/validation_reports/vp/2vpy ftp://data.pdbj.org/pub/pdb/validation_reports/vp/2vpy
ftp://data.pdbj.org/pub/pdb/validation_reports/vp/2vpy リンク
リンク 集合体
集合体
 要素
要素
 THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA4
THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA4
 THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA5
THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA5
 THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA6
THERMUS THERMOPHILUS (バクテリア) / 株: HB27 / 参照: UniProt: Q72LA6







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 23-ID-B / 波長: 0.9333
/ ビームライン: 23-ID-B / 波長: 0.9333  解析
解析 多波長異常分散
多波長異常分散 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj













