[English] 日本語
 Yorodumi
Yorodumi- PDB-2v5z: Structure of human MAO B in complex with the selective inhibitor ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2v5z | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
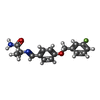
| Title | Structure of human MAO B in complex with the selective inhibitor safinamide | |||||||||
 Components Components | Amine oxidase [flavin-containing] B | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / FAD / MEMBRANE / SAFINAMIDE / FLAVOPROTEIN / HUMAN MAO B STRUCTURE / REVERSIBLE INHIBITOR BINDING / MITOCHONDRION / TRANSMEMBRANE / NEUROPROTECTION / PARKINSON'S DISEASE | |||||||||
| Function / homology |  Function and homology information Function and homology informationBiogenic amines are oxidatively deaminated to aldehydes by MAOA and MAOB / monoamine oxidase / monoamine oxidase activity / primary-amine oxidase / primary methylamine oxidase activity / dopamine catabolic process / mitochondrial envelope / hydrogen peroxide biosynthetic process / substantia nigra development / flavin adenine dinucleotide binding ...Biogenic amines are oxidatively deaminated to aldehydes by MAOA and MAOB / monoamine oxidase / monoamine oxidase activity / primary-amine oxidase / primary methylamine oxidase activity / dopamine catabolic process / mitochondrial envelope / hydrogen peroxide biosynthetic process / substantia nigra development / flavin adenine dinucleotide binding / mitochondrial outer membrane / electron transfer activity / mitochondrion Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.6 Å MOLECULAR REPLACEMENT / Resolution: 1.6 Å | |||||||||
 Authors Authors | Binda, C. / Wang, J. / Pisani, L. / Caccia, C. / Carotti, A. / Salvati, P. / Edmondson, D.E. / Mattevi, A. | |||||||||
 Citation Citation |  Journal: J.Med.Chem. / Year: 2007 Journal: J.Med.Chem. / Year: 2007Title: Structures of human monoamine oxidase B complexes with selective noncovalent inhibitors: safinamide and coumarin analogs. Authors: Binda, C. / Wang, J. / Pisani, L. / Caccia, C. / Carotti, A. / Salvati, P. / Edmondson, D.E. / Mattevi, A. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2v5z.cif.gz 2v5z.cif.gz | 224.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2v5z.ent.gz pdb2v5z.ent.gz | 179.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2v5z.json.gz 2v5z.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/v5/2v5z https://data.pdbj.org/pub/pdb/validation_reports/v5/2v5z ftp://data.pdbj.org/pub/pdb/validation_reports/v5/2v5z ftp://data.pdbj.org/pub/pdb/validation_reports/v5/2v5z | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2v60C  2v61C  1oj9S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments: Ens-ID: 1 / Refine code: 1
NCS oper: (Code: given Matrix: (-0.52735, -0.49561, -0.69012), Vector: |
- Components
Components
| #1: Protein | Mass: 58837.730 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: MAOB / Production host: Homo sapiens (human) / Gene: MAOB / Production host:  PICHIA PASTORIS (fungus) / References: UniProt: P27338, monoamine oxidase PICHIA PASTORIS (fungus) / References: UniProt: P27338, monoamine oxidase#2: Chemical | #3: Chemical | #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.61 Å3/Da / Density % sol: 52.51 % / Description: NONE |
|---|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID14-2 / Wavelength: 0.97 / Beamline: ID14-2 / Wavelength: 0.97 |
| Detector | Type: ADSC CCD / Detector: CCD / Date: Jul 12, 2007 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97 Å / Relative weight: 1 |
| Reflection | Resolution: 1.6→30 Å / Num. obs: 164454 / % possible obs: 97.9 % / Redundancy: 3.1 % / Rmerge(I) obs: 0.08 |
| Reflection shell | Resolution: 1.6→1.64 Å / % possible obs: 98.5 % / Redundancy: 2.9 % / Mean I/σ(I) obs: 1.5 / Num. unique obs: 11773 / Rsym value: 0.466 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 1OJ9 Resolution: 1.6→15 Å / Cor.coef. Fo:Fc: 0.945 / Cor.coef. Fo:Fc free: 0.931 / SU B: 1.598 / SU ML: 0.057 / Cross valid method: THROUGHOUT / ESU R: 0.09 / ESU R Free: 0.087 / Stereochemistry target values: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 15.765 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 1.6→15 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller













































 PDBj
PDBj