| 登録情報 | データベース: PDB / ID: 2n71
|
|---|
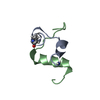
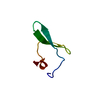
| タイトル | NMR structure of CmPI-II, a serin protease inhibitor isolated from mollusk Cenchitis muricatus |
|---|
 要素 要素 | Protease inhibitor 2 |
|---|
 キーワード キーワード | HYDROLASE INHIBITOR / non-classical Kazal-inhibitor |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
serine-type endopeptidase inhibitor activity類似検索 - 分子機能 Kazal-type serine protease inhibitor domain / Wheat Germ Agglutinin (Isolectin 2); domain 1 - #30 / Kazal type serine protease inhibitors / Kazal domain superfamily / Kazal domain / Kazal domain profile. / Wheat Germ Agglutinin (Isolectin 2); domain 1 / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Cenchritis muricatus (無脊椎動物) Cenchritis muricatus (無脊椎動物) |
|---|
| 手法 | 溶液NMR / simulated annealing, torsion angle dynamics |
|---|
| Model details | lowest energy, model1 |
|---|
 データ登録者 データ登録者 | Cabrera-Munoz, A. / Rojas, L. / Alonso del Rivero Antigua, M. / Pires, J. |
|---|
 引用 引用 |  ジャーナル: J.Struct.Biol. / 年: 2019 ジャーナル: J.Struct.Biol. / 年: 2019
タイトル: NMR structure of CmPI-II, a non-classical Kazal protease inhibitor: Understanding its conformational dynamics and subtilisin A inhibition.
著者: Cabrera-Munoz, A. / Valiente, P.A. / Rojas, L. / Alonso-Del-Rivero Antigua, M. / Pires, J.R. |
|---|
| 履歴 | | 登録 | 2015年9月1日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年9月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年10月21日 | Group: Data collection / Database references
カテゴリ: citation / citation_author ...citation / citation_author / pdbx_nmr_software / pdbx_nmr_spectrometer
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.3 | 2024年11月20日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Cenchritis muricatus (無脊椎動物)
Cenchritis muricatus (無脊椎動物) データ登録者
データ登録者 引用
引用 ジャーナル: J.Struct.Biol. / 年: 2019
ジャーナル: J.Struct.Biol. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2n71.cif.gz
2n71.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2n71.ent.gz
pdb2n71.ent.gz PDB形式
PDB形式 2n71.json.gz
2n71.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2n71_validation.pdf.gz
2n71_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2n71_full_validation.pdf.gz
2n71_full_validation.pdf.gz 2n71_validation.xml.gz
2n71_validation.xml.gz 2n71_validation.cif.gz
2n71_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n7/2n71
https://data.pdbj.org/pub/pdb/validation_reports/n7/2n71 ftp://data.pdbj.org/pub/pdb/validation_reports/n7/2n71
ftp://data.pdbj.org/pub/pdb/validation_reports/n7/2n71 リンク
リンク 集合体
集合体
 要素
要素 Cenchritis muricatus (無脊椎動物)
Cenchritis muricatus (無脊椎動物) Pichia pastoris (菌類) / 参照: UniProt: P84755
Pichia pastoris (菌類) / 参照: UniProt: P84755 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
 HSQC
HSQC