| 登録情報 | データベース: PDB / ID: 2n5k
|
|---|
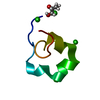
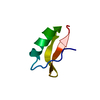
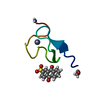
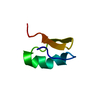
| タイトル | Regnase-1 Zinc finger domain |
|---|
 要素 要素 | Ribonuclease ZC3H12A |
|---|
 キーワード キーワード | HYDROLASE / Regnase / Regnase-1 / Zc3h12a / Zinc finger |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
immune response-activating signaling pathway / positive regulation of miRNA catabolic process / positive regulation of protein deubiquitination / negative regulation of muscle cell apoptotic process / RNA exonuclease activity / miRNA catabolic process / negative regulation of T-helper 17 cell differentiation / rough endoplasmic reticulum membrane / positive regulation of mRNA catabolic process / host-mediated suppression of viral genome replication ...immune response-activating signaling pathway / positive regulation of miRNA catabolic process / positive regulation of protein deubiquitination / negative regulation of muscle cell apoptotic process / RNA exonuclease activity / miRNA catabolic process / negative regulation of T-helper 17 cell differentiation / rough endoplasmic reticulum membrane / positive regulation of mRNA catabolic process / host-mediated suppression of viral genome replication / cellular response to sodium arsenite / negative regulation of cardiac muscle contraction / cellular response to ionomycin / 3'-UTR-mediated mRNA destabilization / nuclease activity / negative regulation of nitric oxide biosynthetic process / positive regulation of lipid storage / cellular response to chemokine / mRNA 3'-UTR AU-rich region binding / positive regulation of p38MAPK cascade / negative regulation of non-canonical NF-kappaB signal transduction / miRNA binding / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / negative regulation of interleukin-1 beta production / mRNA catabolic process / negative regulation of protein phosphorylation / protein complex oligomerization / negative regulation of interleukin-6 production / negative regulation of type II interferon production / negative regulation of tumor necrosis factor production / cellular response to interleukin-1 / positive regulation of execution phase of apoptosis / protein deubiquitination / positive regulation of fat cell differentiation / cellular response to glucose starvation / rough endoplasmic reticulum / positive regulation of defense response to virus by host / negative regulation of canonical NF-kappaB signal transduction / positive regulation of autophagy / negative regulation of cytokine production involved in inflammatory response / RNA endonuclease activity / positive regulation of endothelial cell migration / mRNA 3'-UTR binding / P-body / cellular response to virus / positive regulation of protein import into nucleus / RNA stem-loop binding / positive regulation of reactive oxygen species metabolic process / positive regulation of angiogenesis / cellular response to tumor necrosis factor / nervous system development / T cell receptor signaling pathway / ribosome binding / cellular response to lipopolysaccharide / cellular response to oxidative stress / angiogenesis / 加水分解酵素; エステル加水分解酵素 / cysteine-type deubiquitinase activity / cell differentiation / cytoskeleton / inflammatory response / negative regulation of gene expression / mRNA binding / apoptotic process / DNA damage response / chromatin binding / positive regulation of gene expression / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm類似検索 - 分子機能 Rege-1, UBA-like domain / Endoribonuclease Regnase 1/ZC3H12, C-terminal domain / UBA-like domain / Endoribonuclease Regnase 1/ ZC3H12 C-terminal domain / Ribonuclease Zc3h12a-like, NYN domain / : / Zc3h12a-like Ribonuclease NYN domain類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 | 溶液NMR / distance geometry, torsion angle dynamics |
|---|
| Model details | lowest energy, model1 |
|---|
 データ登録者 データ登録者 | Yokogawa, M. / Tsushima, T. / Noda, N.N. / Kumeta, H. / Adachi, W. / Enokizono, Y. / Yamashita, K. / Standley, D.M. / Takeuchi, O. / Akira, S. / Inagaki, F. |
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2016 ジャーナル: Sci Rep / 年: 2016
タイトル: Structural basis for the regulation of enzymatic activity of Regnase-1 by domain-domain interactions
著者: Yokogawa, M. / Tsushima, T. / Noda, N.N. / Kumeta, H. / Enokizono, Y. / Yamashita, K. / Standley, D.M. / Takeuchi, O. / Akira, S. / Inagaki, F. |
|---|
| 履歴 | | 登録 | 2015年7月18日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2016年3月16日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年6月14日 | Group: Database references / Derived calculations / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_struct_conn_angle / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.2 | 2024年10月30日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: Sci Rep / 年: 2016
ジャーナル: Sci Rep / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2n5k.cif.gz
2n5k.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2n5k.ent.gz
pdb2n5k.ent.gz PDB形式
PDB形式 2n5k.json.gz
2n5k.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2n5k_validation.pdf.gz
2n5k_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2n5k_full_validation.pdf.gz
2n5k_full_validation.pdf.gz 2n5k_validation.xml.gz
2n5k_validation.xml.gz 2n5k_validation.cif.gz
2n5k_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n5/2n5k
https://data.pdbj.org/pub/pdb/validation_reports/n5/2n5k ftp://data.pdbj.org/pub/pdb/validation_reports/n5/2n5k
ftp://data.pdbj.org/pub/pdb/validation_reports/n5/2n5k リンク
リンク 集合体
集合体
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


 HSQC
HSQC