+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2ml2 | ||||||
|---|---|---|---|---|---|---|---|
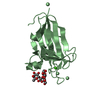
| タイトル | Solution Structure of AlgE6R2 subunit from the Azotobacter vinelandii Mannuronan C5-epimerase | ||||||
 要素 要素 | Poly(beta-D-mannuronate) C5 epimerase 6 | ||||||
 キーワード キーワード | ISOMERASE / Alginate C-5 epimerase / mannuronan C-5 epimerase / R-module | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mannuronan 5-epimerase / alginic acid biosynthetic process / isomerase activity / calcium ion binding / extracellular space 類似検索 - 分子機能 | ||||||
| 生物種 |  Azotobacter vinelandii (窒素固定) Azotobacter vinelandii (窒素固定) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
| Model details | lowest energy, model1 | ||||||
 データ登録者 データ登録者 | Buchinger, E. / Wimmer, R. / Aachmann, F.L. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2014 ジャーナル: J.Biol.Chem. / 年: 2014タイトル: Structural and Functional Characterization of the R-modules in Alginate C-5 Epimerases AlgE4 and AlgE6 from Azotobacter vinelandii 著者: Buchinger, E. / Knudsen, D.H. / Behrens, M.A. / Pedersen, J.S. / Aarstad, O.A. / Tndervik, A. / Valla, S. / Skjak-Brk, G. / Wimmer, R. / Aachmann, F.L. #1: ジャーナル: Biomol.Nmr Assign. / 年: 2011 タイトル: 1H, 13C and 15N resonances of the AlgE62 subunit from Azotobacter vinelandii mannuronan C5-epimerase 著者: Andreassen, T. / Buchinger, E. / Skjak-Brk, G. / Valla, S. / Aachmann, F.L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2ml2.cif.gz 2ml2.cif.gz | 893.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2ml2.ent.gz pdb2ml2.ent.gz | 746.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2ml2.json.gz 2ml2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ml/2ml2 https://data.pdbj.org/pub/pdb/validation_reports/ml/2ml2 ftp://data.pdbj.org/pub/pdb/validation_reports/ml/2ml2 ftp://data.pdbj.org/pub/pdb/validation_reports/ml/2ml2 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  2ml1C  2ml3C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 16547.721 Da / 分子数: 1 / 断片: UNP residues 534-694 / 変異: H72Q / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii (窒素固定) Azotobacter vinelandii (窒素固定)株: 354 Eubacteria / 遺伝子: algE6 / 発現宿主:  参照: UniProt: Q9ZFH0, 異性化酵素; ラセマーゼ・エピメラーゼ(光学異性の転換); 炭水化物およびその類縁体に作用 |
|---|---|
| #2: 化合物 | ChemComp-CA / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR 詳細: Structure of the second R-module of AlgE6 from Azotobacter vinelandii | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 試料状態 | イオン強度: 0.165 / pH: 6.9 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker Avance / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||||||||||||||||||
| NMR constraints | NOE constraints total: 3579 / NOE intraresidue total count: 2316 / NOE long range total count: 397 / NOE medium range total count: 106 / NOE sequential total count: 760 / Protein chi angle constraints total count: 0 / Protein other angle constraints total count: 0 / Protein phi angle constraints total count: 52 / Protein psi angle constraints total count: 52 | ||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 / 代表コンフォーマー: 1 |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj

 HSQC
HSQC