+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2kjw | ||||||
|---|---|---|---|---|---|---|---|
| Title | Solution structure and backbone dynamics of the permutant P54-55 | ||||||
 Components Components | 30S ribosomal protein S6 | ||||||
 Keywords Keywords | RIBOSOMAL PROTEIN / S6 permutant / solution structure / backbone dynamics / folding / Ribonucleoprotein / RNA-binding / rRNA-binding | ||||||
| Function / homology |  Function and homology information Function and homology informationsmall ribosomal subunit rRNA binding / structural constituent of ribosome / ribosome / translation / ribonucleoprotein complex / cytoplasm Similarity search - Function | ||||||
| Biological species |   Thermus thermophilus (bacteria) Thermus thermophilus (bacteria) | ||||||
| Method | SOLUTION NMR / simulated annealing | ||||||
| Model details | lowest energy, model 1 | ||||||
 Authors Authors | Ohman, A. / Oliveberg, M. / Oman, T. | ||||||
 Citation Citation |  Journal: Protein Sci. / Year: 2010 Journal: Protein Sci. / Year: 2010Title: Solution structures and backbone dynamics of the ribosomal protein S6 and its permutant P(54-55) Authors: Ohman, A. / Oman, T. / Oliveberg, M. #1: Journal: Proc.Natl.Acad.Sci.USA / Year: 2009 Title: The HD-exchange motions of ribosomal protein S6 are insensitive to reversal of the protein-folding pathway Authors: Haglund, E. / Lind, J. / Oman, T. / Ohman, A. / Maler, L. / Oliveberg, M. | ||||||
| History |
|
- Structure visualization
Structure visualization
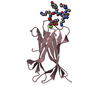
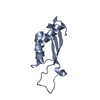
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2kjw.cif.gz 2kjw.cif.gz | 620.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2kjw.ent.gz pdb2kjw.ent.gz | 519.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2kjw.json.gz 2kjw.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/kj/2kjw https://data.pdbj.org/pub/pdb/validation_reports/kj/2kjw ftp://data.pdbj.org/pub/pdb/validation_reports/kj/2kjw ftp://data.pdbj.org/pub/pdb/validation_reports/kj/2kjw | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2kjvC C: citing same article ( |
|---|---|
| Similar structure data | |
| Other databases |
|
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: Protein | Mass: 11258.886 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Details: Permutant of the ribosomal protein S6 from Thermus thermophilus Source: (gene. exp.)   Thermus thermophilus (bacteria) / Gene: rpsF, rps6 / Production host: Thermus thermophilus (bacteria) / Gene: rpsF, rps6 / Production host:  |
|---|
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR Details: Solution structure and backbone dynamics of the permutant S6p3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
|
- Sample preparation
Sample preparation
| Details |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Sample |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Sample conditions | pH: 6.3 / Pressure: ambient / Temperature: 298 K |
-NMR measurement
| NMR spectrometer | Type: Bruker DRX / Manufacturer: Bruker / Model: DRX / Field strength: 600 MHz |
|---|
- Processing
Processing
| NMR software |
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method: simulated annealing / Software ordinal: 1 Details: sa.inp and refine.inp used with slight modifications. | ||||||||||||||||||||||||||||||||||||||||||
| NMR constraints | NOE constraints total: 770 / NOE intraresidue total count: 213 / NOE long range total count: 116 / NOE medium range total count: 138 / NOE sequential total count: 303 / Protein phi angle constraints total count: 88 / Protein psi angle constraints total count: 65 | ||||||||||||||||||||||||||||||||||||||||||
| NMR representative | Selection criteria: lowest energy | ||||||||||||||||||||||||||||||||||||||||||
| NMR ensemble | Conformer selection criteria: structures with the least restraint violations Conformers calculated total number: 200 / Conformers submitted total number: 20 |
 Movie
Movie Controller
Controller












 PDBj
PDBj




 HSQC
HSQC