| 登録情報 | データベース: PDB / ID: 2jx9
|
|---|
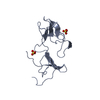
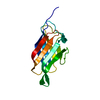
| タイトル | Solution structure of the Gal_lectin domain of mouse Latrophilin-1 GPCR |
|---|
 要素 要素 | Latrophilin 1 |
|---|
 キーワード キーワード | CELL ADHESION / SIGNALING PROTEIN / Lectin / beta-sandwich / disulphide / glycosylated / G-protein coupled receptor / Membrane / Receptor / Transducer / Transmembrane |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
latrotoxin receptor activity / positive regulation of synapse assembly / cell adhesion molecule binding / G protein-coupled receptor activity / presynaptic membrane / growth cone / carbohydrate binding / cell surface receptor signaling pathway / neuron projection / axon ...latrotoxin receptor activity / positive regulation of synapse assembly / cell adhesion molecule binding / G protein-coupled receptor activity / presynaptic membrane / growth cone / carbohydrate binding / cell surface receptor signaling pathway / neuron projection / axon / synapse / plasma membrane類似検索 - 分子機能 Latrophilin-1 / Rhamnose-binding lectin domain (RBL) / GPCR, family 2, latrophilin, C-terminal / GPCR, family 2, latrophilin / Latrophilin Cytoplasmic C-terminal region / D-galactoside/L-rhamnose binding SUEL lectin domain superfamily / D-galactoside/L-rhamnose binding SUEL lectin domain / D-galactoside/L-rhamnose binding SUEL lectin domain / SUEL-type lectin domain profile. / GAIN domain, N-terminal ...Latrophilin-1 / Rhamnose-binding lectin domain (RBL) / GPCR, family 2, latrophilin, C-terminal / GPCR, family 2, latrophilin / Latrophilin Cytoplasmic C-terminal region / D-galactoside/L-rhamnose binding SUEL lectin domain superfamily / D-galactoside/L-rhamnose binding SUEL lectin domain / D-galactoside/L-rhamnose binding SUEL lectin domain / SUEL-type lectin domain profile. / GAIN domain, N-terminal / GPCR-Autoproteolysis INducing (GAIN) domain / Olfactomedin-like domain / Olfactomedin-like domain / Olfactomedin-like domain profile. / Olfactomedin-like domains / GAIN domain superfamily / GPCR proteolysis site, GPS, motif / GPS motif / GAIN-B domain profile. / G-protein-coupled receptor proteolytic site domain / GPCR, family 2, extracellular hormone receptor domain / G-protein coupled receptors family 2 profile 1. / Domain present in hormone receptors / Hormone receptor domain / GPCR family 2, extracellular hormone receptor domain superfamily / G-protein coupled receptors family 2 signature 2. / GPCR, family 2, secretin-like, conserved site / GPCR, family 2, secretin-like / 7 transmembrane receptor (Secretin family) / GPCR, family 2-like / G-protein coupled receptors family 2 profile 2. / Jelly Rolls / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 | 溶液NMR / torsion angle dynamics, simulated annealing |
|---|
 データ登録者 データ登録者 | Vakonakis, I. / Campbell, I.D. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: Solution structure and sugar-binding mechanism of mouse Latrophilin-1 RBL: a novel 7TM receptor-attached lectin-like domain.
著者: Vakonakis, I. / Langenhan, T. / Promel, S. / Russ, A. / Campbell, I.D. |
|---|
| 履歴 | | 登録 | 2007年11月9日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2008年4月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Non-polymer description / Version format compliance |
|---|
| 改定 1.2 | 2020年2月26日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other
カテゴリ: chem_comp / database_2 ...chem_comp / database_2 / pdbx_database_status / pdbx_nmr_software / struct_conn
Item: _chem_comp.type / _pdbx_database_status.status_code_cs ..._chem_comp.type / _pdbx_database_status.status_code_cs / _pdbx_nmr_software.name / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 1.3 | 2020年7月29日 | Group: Data collection / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_conn / struct_site / struct_site_gen
Item: _chem_comp.name / _entity.pdbx_description ..._chem_comp.name / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_conn.pdbx_role
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.4 | 2023年6月14日 | Group: Database references / Other / Structure summary / カテゴリ: chem_comp / database_2 / pdbx_database_status
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.5 | 2024年10月16日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2jx9.cif.gz
2jx9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2jx9.ent.gz
pdb2jx9.ent.gz PDB形式
PDB形式 2jx9.json.gz
2jx9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2jx9_validation.pdf.gz
2jx9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2jx9_full_validation.pdf.gz
2jx9_full_validation.pdf.gz 2jx9_validation.xml.gz
2jx9_validation.xml.gz 2jx9_validation.cif.gz
2jx9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/jx/2jx9
https://data.pdbj.org/pub/pdb/validation_reports/jx/2jx9 ftp://data.pdbj.org/pub/pdb/validation_reports/jx/2jx9
ftp://data.pdbj.org/pub/pdb/validation_reports/jx/2jx9
 リンク
リンク 集合体
集合体
 要素
要素
 Pichia pastoris (菌類) / 株 (発現宿主): X-33 / 参照: UniProt: Q5U4D5, UniProt: Q80TR1*PLUS
Pichia pastoris (菌類) / 株 (発現宿主): X-33 / 参照: UniProt: Q5U4D5, UniProt: Q80TR1*PLUS 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


 HSQC
HSQC