登録情報 データベース : PDB / ID : 2iuhタイトル Crystal structure of the PI3-kinase p85 N-terminal SH2 domain in complex with c-Kit phosphotyrosyl peptide C-KIT PHOSPHOTYROSYL PEPTIDE PHOSPHATIDYLINOSITOL 3-KINASE REGULATORY ALPHA SUBUNIT キーワード / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)手法 / OTHER / 解像度 : 2 Å データ登録者 Nolte, R.T. / Eck, M.J. / Schlessinger, J. / Shoelson, S.E. / Harrison, S.C. ジャーナル : Nat.Struct.Biol. / 年 : 1996タイトル : Crystal Structure of the Pi 3-Kinase P85 Amino-Terminal Sh2 Domain and its Phosphopeptide Complexes著者 : Nolte, R.T. / Eck, M.J. / Schlessinger, J. / Shoelson, S.E. / Harrison, S.C. 履歴 登録 2006年6月3日 登録サイト / 処理サイト 改定 1.0 2006年6月6日 Provider / タイプ 改定 1.1 2015年10月28日 Group Database references / Derived calculations ... Database references / Derived calculations / Non-polymer description / Other / Source and taxonomy / Structure summary / Version format compliance 改定 1.2 2021年4月28日 Group / Derived calculations / カテゴリ / struct_connItem / _struct_conn.pdbx_leaving_atom_flag改定 1.3 2024年11月20日 Group Data collection / Database references ... Data collection / Database references / Refinement description / Structure summary カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / refine / reflns Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_entry_details.has_protein_modification / _refine.ls_number_reflns_obs / _reflns.observed_criterion_sigma_I
すべて表示 表示を減らす Remark 700 SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN ... SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN THE SHEET RECORDS BELOW, TWO SHEETS ARE DEFINED.
 データを開く
データを開く 基本情報
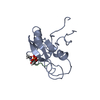
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 / OTHER / 解像度: 2 Å
X線回折 / OTHER / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Biol. / 年: 1996
ジャーナル: Nat.Struct.Biol. / 年: 1996 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2iuh.cif.gz
2iuh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2iuh.ent.gz
pdb2iuh.ent.gz PDB形式
PDB形式 2iuh.json.gz
2iuh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2iuh_validation.pdf.gz
2iuh_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2iuh_full_validation.pdf.gz
2iuh_full_validation.pdf.gz 2iuh_validation.xml.gz
2iuh_validation.xml.gz 2iuh_validation.cif.gz
2iuh_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/iu/2iuh
https://data.pdbj.org/pub/pdb/validation_reports/iu/2iuh ftp://data.pdbj.org/pub/pdb/validation_reports/iu/2iuh
ftp://data.pdbj.org/pub/pdb/validation_reports/iu/2iuh リンク
リンク 集合体
集合体
 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PET / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PET / 発現宿主: 
 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 X線回折
X線回折 試料調製
試料調製 回転陽極 / 波長: 1.5418
回転陽極 / 波長: 1.5418  解析
解析 X-PLOR / 分類: 精密化
X-PLOR / 分類: 精密化 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj























