[English] 日本語
 Yorodumi
Yorodumi- PDB-2gwa: Crystal Structure of a Complex Formed Between the DNA Holliday Ju... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2gwa | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of a Complex Formed Between the DNA Holliday Junction and a Bis-Acridine Molecule. | ||||||||||||||||||||
 Components Components | 5'-D(* Keywords KeywordsDNA / DNA HOLLIDAY JUNCTION BIS-ACRIDINE | Function / homology | Chem-A4C / SPERMINE / DNA |  Function and homology information Function and homology informationMethod |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / Resolution: 1.75 Å SYNCHROTRON / Resolution: 1.75 Å  Authors AuthorsBrogden, A.L. / Hopcroft, N.H. / Cardin, C.J. / Searcey, M. |  Citation Citation Journal: Angew.Chem.Int.Ed.Engl. / Year: 2007 Journal: Angew.Chem.Int.Ed.Engl. / Year: 2007Title: Ligand bridging of the DNA Holliday junction: molecular recognition of a stacked-X four-way junction by a small molecule. Authors: Brogden, A.L. / Hopcroft, N.H. / Searcey, M. / Cardin, C.J. History |
Remark 600 | HETEROGEN HETEROGEN A4C 1 REPRESENTS ONE HALF OF THE LIGAND, THE OTHER HALF IS GENERATED BY THE ...HETEROGEN HETEROGEN A4C 1 REPRESENTS ONE HALF OF THE LIGAND, THE OTHER HALF IS GENERATED BY THE SYMMETRY OPERATION USED TO GENERATE THE BIOLOGICAL UNIT. THE MOLECULE AS A WHOLE LIES ON A CRYSTALLOGRAPHIC AXIS OF TWO-FOLD ROTATIONAL SYMMETRY. HENCE, IN THE ASYMMETRIC UNIT, ONLY HALF THE LIGAND IS PRESENT IN THE COORDINATE FILE SUBMITTED. LINKS ARE PROVIDED BETWEEN SYMMETRY RELATED CX6 ATOMS. | |
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2gwa.cif.gz 2gwa.cif.gz | 28.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2gwa.ent.gz pdb2gwa.ent.gz | 16.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2gwa.json.gz 2gwa.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/gw/2gwa https://data.pdbj.org/pub/pdb/validation_reports/gw/2gwa ftp://data.pdbj.org/pub/pdb/validation_reports/gw/2gwa ftp://data.pdbj.org/pub/pdb/validation_reports/gw/2gwa | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1nqsS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 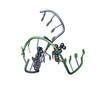
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
| |||||||||
| Details | THE SECOND PART OF THE BIOLOGICAL UNIT IS GENERATED BY THE SYMMETRY OPERATION: -X, Y, -Z. |
- Components
Components
| #1: DNA chain | Mass: 3045.005 Da / Num. of mol.: 2 / Source method: obtained synthetically #2: Chemical | ChemComp-A4C / | #3: Chemical | ChemComp-SPM / | #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.26 Å3/Da / Density % sol: 45.69 % | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, sitting drop / pH: 7 Details: 40mM SODIUM CACODYLATE PH 7.0, 12mM SPERMINE, 10% MPD, WITH 35% MPD RESERVOIR., VAPOR DIFFUSION, SITTING DROP, temperature 291K | ||||||||||||||||||||||||||||
| Components of the solutions |
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / Beamline: X11 / Wavelength: 0.806 Å / Beamline: X11 / Wavelength: 0.806 Å |
| Detector | Type: MARRESEARCH / Detector: CCD / Date: Feb 10, 2005 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.806 Å / Relative weight: 1 |
| Reflection | Resolution: 1.75→6 Å / Num. all: 4960 / Num. obs: 4045 / % possible obs: 86.4 % / Observed criterion σ(F): 5 / Observed criterion σ(I): 5 / Redundancy: 2.2 % / Biso Wilson estimate: 23.7 Å2 / Rmerge(I) obs: 0.042 / Net I/σ(I): 13.1 |
| Reflection shell | Resolution: 1.75→1.79 Å / Redundancy: 2.1 % / Rmerge(I) obs: 0.093 / Mean I/σ(I) obs: 5.2 / Num. unique all: 4960 / % possible all: 86.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Starting model: PDB ENTRY 1NQS Resolution: 1.75→6 Å / Num. parameters: 2292 / Num. restraintsaints: 1970 / Cross valid method: FREE R / σ(F): 0 / Stereochemistry target values: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 0 / Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 572 | |||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.75→6 Å
| |||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj