| 登録情報 | データベース: PDB / ID: 2fk2
|
|---|
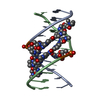
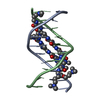
| タイトル | Structure of the Alzheimer's Amyloid Precursor Protein (APP) Copper Binding Domain in 'small unit cell' form, Cu(I)-bound |
|---|
 要素 要素 | Amyloid beta A4 protein precursor |
|---|
 キーワード キーワード | APOPTOSIS / Alpha-Beta Two-layered Sandwich / Cu(I) coordination |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
amyloid-beta complex / growth cone lamellipodium / cellular response to norepinephrine stimulus / growth cone filopodium / microglia development / collateral sprouting in absence of injury / Formyl peptide receptors bind formyl peptides and many other ligands / axo-dendritic transport / regulation of Wnt signaling pathway / regulation of synapse structure or activity ...amyloid-beta complex / growth cone lamellipodium / cellular response to norepinephrine stimulus / growth cone filopodium / microglia development / collateral sprouting in absence of injury / Formyl peptide receptors bind formyl peptides and many other ligands / axo-dendritic transport / regulation of Wnt signaling pathway / regulation of synapse structure or activity / axon midline choice point recognition / astrocyte activation involved in immune response / NMDA selective glutamate receptor signaling pathway / regulation of spontaneous synaptic transmission / mating behavior / growth factor receptor binding / peptidase activator activity / Golgi-associated vesicle / PTB domain binding / positive regulation of amyloid fibril formation / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / Lysosome Vesicle Biogenesis / astrocyte projection / neuron remodeling / Deregulated CDK5 triggers multiple neurodegenerative pathways in Alzheimer's disease models / nuclear envelope lumen / dendrite development / positive regulation of protein metabolic process / TRAF6 mediated NF-kB activation / Advanced glycosylation endproduct receptor signaling / signaling receptor activator activity / negative regulation of long-term synaptic potentiation / modulation of excitatory postsynaptic potential / The NLRP3 inflammasome / transition metal ion binding / main axon / regulation of multicellular organism growth / intracellular copper ion homeostasis / regulation of presynapse assembly / ECM proteoglycans / positive regulation of T cell migration / neuronal dense core vesicle / Purinergic signaling in leishmaniasis infection / positive regulation of chemokine production / cellular response to manganese ion / Notch signaling pathway / clathrin-coated pit / extracellular matrix organization / neuron projection maintenance / Mitochondrial protein degradation / astrocyte activation / ionotropic glutamate receptor signaling pathway / positive regulation of calcium-mediated signaling / positive regulation of mitotic cell cycle / response to interleukin-1 / axonogenesis / protein serine/threonine kinase binding / cellular response to copper ion / platelet alpha granule lumen / cellular response to cAMP / positive regulation of glycolytic process / central nervous system development / positive regulation of interleukin-1 beta production / endosome lumen / dendritic shaft / trans-Golgi network membrane / adult locomotory behavior / positive regulation of long-term synaptic potentiation / learning / positive regulation of JNK cascade / Post-translational protein phosphorylation / locomotory behavior / microglial cell activation / serine-type endopeptidase inhibitor activity / positive regulation of non-canonical NF-kappaB signal transduction / TAK1-dependent IKK and NF-kappa-B activation / regulation of long-term neuronal synaptic plasticity / cellular response to nerve growth factor stimulus / synapse organization / recycling endosome / visual learning / positive regulation of interleukin-6 production / response to lead ion / Golgi lumen / cognition / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / endocytosis / cellular response to amyloid-beta / positive regulation of inflammatory response / neuron projection development / positive regulation of tumor necrosis factor production / Platelet degranulation / heparin binding / regulation of translation / regulation of gene expression / early endosome membrane / perikaryon / G alpha (i) signalling events / G alpha (q) signalling events / dendritic spine類似検索 - 分子機能 Amyloidogenic glycoprotein, copper-binding domain / Amyloidogenic glycoprotein, copper-binding / Amyloidogenic glycoprotein, copper-binding domain conserved site / Amyloidogenic glycoprotein, copper-binding domain superfamily / Copper-binding of amyloid precursor, CuBD / Amyloid precursor protein (APP) copper-binding (CuBD) domain signature. / Amyloidogenic glycoprotein, amyloid-beta peptide superfamily / Beta-amyloid peptide (beta-APP) / Amyloidogenic glycoprotein, amyloid-beta peptide / Beta-amyloid precursor protein C-terminal ...Amyloidogenic glycoprotein, copper-binding domain / Amyloidogenic glycoprotein, copper-binding / Amyloidogenic glycoprotein, copper-binding domain conserved site / Amyloidogenic glycoprotein, copper-binding domain superfamily / Copper-binding of amyloid precursor, CuBD / Amyloid precursor protein (APP) copper-binding (CuBD) domain signature. / Amyloidogenic glycoprotein, amyloid-beta peptide superfamily / Beta-amyloid peptide (beta-APP) / Amyloidogenic glycoprotein, amyloid-beta peptide / Beta-amyloid precursor protein C-terminal / Amyloidogenic glycoprotein, intracellular domain, conserved site / Beta-amyloid precursor protein C-terminus / Amyloid precursor protein (APP) intracellular domain signature. / Amyloidogenic glycoprotein, extracellular / Amyloidogenic glycoprotein, heparin-binding / Amyloidogenic glycoprotein, E2 domain / E2 domain superfamily / Amyloidogenic glycoprotein, heparin-binding domain superfamily / Amyloid A4 N-terminal heparin-binding / E2 domain of amyloid precursor protein / Amyloid precursor protein (APP) E1 domain profile. / Amyloid precursor protein (APP) E2 domain profile. / amyloid A4 / Amyloidogenic glycoprotein / Proteinase inhibitor I2, Kunitz, conserved site / Pancreatic trypsin inhibitor (Kunitz) family signature. / BPTI/Kunitz family of serine protease inhibitors. / Pancreatic trypsin inhibitor Kunitz domain / Kunitz/Bovine pancreatic trypsin inhibitor domain / Pancreatic trypsin inhibitor (Kunitz) family profile. / Pancreatic trypsin inhibitor Kunitz domain superfamily / Dna Ligase; domain 1 / PH-like domain superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 COPPER (I) ION / Amyloid-beta precursor protein類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.65 Å 分子置換 / 解像度: 1.65 Å |
|---|
 データ登録者 データ登録者 | Kong, G.K.-W. / Parker, M.W. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2007 ジャーナル: J.Mol.Biol. / 年: 2007
タイトル: Structural Studies of the Alzheimer's Amyloid Precursor Protein Copper-binding Domain Reveal How it Binds Copper Ions
著者: Kong, G.K. / Adams, J.J. / Harris, H.H. / Boas, J.F. / Curtain, C.C. / Galatis, D. / Masters, C.L. / Barnham, K.J. / McKinstry, W.J. / Cappai, R. / Parker, M.W. |
|---|
| 履歴 | | 登録 | 2006年1月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2007年1月16日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2023年8月30日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2024年10月16日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.65 Å
分子置換 / 解像度: 1.65 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2007
ジャーナル: J.Mol.Biol. / 年: 2007 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2fk2.cif.gz
2fk2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2fk2.ent.gz
pdb2fk2.ent.gz PDB形式
PDB形式 2fk2.json.gz
2fk2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2fk2_validation.pdf.gz
2fk2_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2fk2_full_validation.pdf.gz
2fk2_full_validation.pdf.gz 2fk2_validation.xml.gz
2fk2_validation.xml.gz 2fk2_validation.cif.gz
2fk2_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/fk/2fk2
https://data.pdbj.org/pub/pdb/validation_reports/fk/2fk2 ftp://data.pdbj.org/pub/pdb/validation_reports/fk/2fk2
ftp://data.pdbj.org/pub/pdb/validation_reports/fk/2fk2 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: APP / プラスミド: pPIC-9 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: APP / プラスミド: pPIC-9 / 発現宿主:  Pichia pastoris (菌類) / 株 (発現宿主): GS115 / 参照: UniProt: P05067
Pichia pastoris (菌類) / 株 (発現宿主): GS115 / 参照: UniProt: P05067 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 14-ID-B / 波長: 1.3783 Å
/ ビームライン: 14-ID-B / 波長: 1.3783 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

















