| 登録情報 | データベース: PDB / ID: 2fci
|
|---|
| タイトル | Structural basis for the requirement of two phosphotyrosines in signaling mediated by Syk tyrosine kinase |
|---|
 要素 要素 | - C-termainl SH2 domain from phospholipase C-gamma-1 comprising residues 663-759
- Doubly phosphorylated peptide derived from Syk kinase comprising residues 338-350
|
|---|
 キーワード キーワード | HYDROLASE / SH2 domain / phosphopeptide / Syk kinase / PLCgamma / PLCC |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
calcium-dependent phospholipase C activity / phosphoinositide phospholipase C / phosphatidylinositol metabolic process / COP9 signalosome / phosphatidylinositol-4,5-bisphosphate phospholipase C activity / phospholipid catabolic process / phosphatidylinositol-mediated signaling / positive regulation of epithelial cell migration / immune system process / cellular response to vascular endothelial growth factor stimulus ...calcium-dependent phospholipase C activity / phosphoinositide phospholipase C / phosphatidylinositol metabolic process / COP9 signalosome / phosphatidylinositol-4,5-bisphosphate phospholipase C activity / phospholipid catabolic process / phosphatidylinositol-mediated signaling / positive regulation of epithelial cell migration / immune system process / cellular response to vascular endothelial growth factor stimulus / ruffle / release of sequestered calcium ion into cytosol / cellular response to epidermal growth factor stimulus / guanyl-nucleotide exchange factor activity / non-membrane spanning protein tyrosine kinase activity / non-specific protein-tyrosine kinase / epidermal growth factor receptor signaling pathway / ruffle membrane / lamellipodium / in utero embryonic development / intracellular signal transduction / calcium ion binding / ATP binding / plasma membrane / cytoplasm類似検索 - 分子機能 1-phosphatidylinositol 4,5-bisphosphate phosphodiesterase gamma-1, SH3 domain / PLCG EF-hand motif 1 / Phosphatidylinositol-4, 5-bisphosphate phosphodiesterase gamma / PLC-gamma, C-terminal SH2 domain / PLC-gamma, N-terminal SH2 domain / PLCG EF-hand motif 2 / Phosphoinositide phospholipase C family / Phospholipase C, phosphatidylinositol-specific, Y domain / Phosphatidylinositol-specific phospholipase C, Y domain / Phosphatidylinositol-specific phospholipase Y-box domain profile. ...1-phosphatidylinositol 4,5-bisphosphate phosphodiesterase gamma-1, SH3 domain / PLCG EF-hand motif 1 / Phosphatidylinositol-4, 5-bisphosphate phosphodiesterase gamma / PLC-gamma, C-terminal SH2 domain / PLC-gamma, N-terminal SH2 domain / PLCG EF-hand motif 2 / Phosphoinositide phospholipase C family / Phospholipase C, phosphatidylinositol-specific, Y domain / Phosphatidylinositol-specific phospholipase C, Y domain / Phosphatidylinositol-specific phospholipase Y-box domain profile. / Phospholipase C, catalytic domain (part); domain Y / Tyrosine-protein kinase, non-receptor SYK/ZAP-70 / Tyrosine-protein kinase SYK/ZAP-70, inter-SH2 domain superfamily / SYK/ZAP-70, N-terminal SH2 domain / Phosphatidylinositol-specific phospholipase C, X domain / Phosphatidylinositol-specific phospholipase C, X domain / Phospholipase C, catalytic domain (part); domain X / Phosphatidylinositol-specific phospholipase X-box domain profile. / PLC-like phosphodiesterase, TIM beta/alpha-barrel domain superfamily / SH2 domain / SHC Adaptor Protein / Protein kinase C conserved region 2 (CalB) / C2 domain / C2 domain / C2 domain profile. / PH domain / : / C2 domain superfamily / PH domain profile. / Pleckstrin homology domain. / Pleckstrin homology domain / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / SH2 domain superfamily / Src homology 3 domains / EF-Hand 1, calcium-binding site / SH3-like domain superfamily / EF-hand calcium-binding domain. / Src homology 3 (SH3) domain profile. / SH3 domain / EF-hand calcium-binding domain profile. / EF-hand domain / EF-hand domain pair / Tyrosine-protein kinase, catalytic domain / Tyrosine kinase, catalytic domain / Tyrosine protein kinases specific active-site signature. / Tyrosine-protein kinase, active site / PH-like domain superfamily / Serine-threonine/tyrosine-protein kinase, catalytic domain / Protein tyrosine and serine/threonine kinase / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 1-phosphatidylinositol 4,5-bisphosphate phosphodiesterase gamma-1 / Tyrosine-protein kinase類似検索 - 構成要素 |
|---|
| 生物種 |   Bos taurus (ウシ) Bos taurus (ウシ) |
|---|
| 手法 | 溶液NMR |
|---|
| Model type details | minimized average |
|---|
 データ登録者 データ登録者 | Groesch, T.D. / Zhou, F. / Mattila, S. / Geahlen, R.L. / Post, C.B. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2006 ジャーナル: J.Mol.Biol. / 年: 2006
タイトル: Structural basis for the requirement of two phosphotyrosine residues in signaling mediated by syk tyrosine kinase
著者: Groesch, T.D. / Zhou, F. / Mattila, S. / Geahlen, R.L. / Post, C.B. |
|---|
| 履歴 | | 登録 | 2005年12月12日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2006年1月31日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2012年5月2日 | Group: Structure summary |
|---|
| 改定 2.0 | 2023年11月15日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / database_2 / pdbx_validate_close_contact / struct_conn / struct_ref_seq_dif
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_validate_close_contact.auth_atom_id_1 / _pdbx_validate_close_contact.auth_atom_id_2 / _struct_conn.pdbx_leaving_atom_flag / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2006
ジャーナル: J.Mol.Biol. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2fci.cif.gz
2fci.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2fci.ent.gz
pdb2fci.ent.gz PDB形式
PDB形式 2fci.json.gz
2fci.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/fc/2fci
https://data.pdbj.org/pub/pdb/validation_reports/fc/2fci ftp://data.pdbj.org/pub/pdb/validation_reports/fc/2fci
ftp://data.pdbj.org/pub/pdb/validation_reports/fc/2fci リンク
リンク 集合体
集合体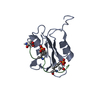
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










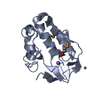


 PDBj
PDBj



 HSQC
HSQC