+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2f43 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Rat liver F1-ATPase | ||||||
 要素 要素 | (ATP synthase ...) x 3 | ||||||
 キーワード キーワード | HYDROLASE / ATP SYNTHASE / F0F1-ATPASE / OXIDATIVE PHOSPHORYLATION / MITOCHONDRIA / VANADATE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lipoprotein particle receptor activity / Mitochondrial protein import / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / negative regulation of cell adhesion involved in substrate-bound cell migration / response to 3,3',5-triiodo-L-thyronine / cold acclimation / response to curcumin / cellular response to peptide ...lipoprotein particle receptor activity / Mitochondrial protein import / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / negative regulation of cell adhesion involved in substrate-bound cell migration / response to 3,3',5-triiodo-L-thyronine / cold acclimation / response to curcumin / cellular response to peptide / angiostatin binding / ATP biosynthetic process / response to manganese ion / cellular response to interleukin-7 / response to muscle activity / negative regulation of endothelial cell proliferation / proton motive force-driven ATP synthesis / MHC class I protein binding / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATPase activity, rotational mechanism / positive regulation of blood vessel endothelial cell migration / cellular response to dexamethasone stimulus / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / cellular response to nitric oxide / proton transmembrane transport / receptor-mediated endocytosis / regulation of intracellular pH / liver development / ADP binding / lipid metabolic process / mitochondrial membrane / protease binding / response to ethanol / angiogenesis / mitochondrial inner membrane / membrane raft / calcium ion binding / cell surface / ATP hydrolysis activity / mitochondrion / ATP binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3 Å 分子置換 / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Chen, C. / Saxena, A.K. / Simcoke, W.N. / Garboczi, D.N. / Pedersen, P.L. / Ko, Y.H. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2006 ジャーナル: J.Biol.Chem. / 年: 2006タイトル: Mitochondrial ATP synthase: Crystal structure of the catalytic F1 unit in a vanadate-induced transition-like state and implications for mechanism. 著者: Chen, C. / Saxena, A.K. / Simcoke, W.N. / Garboczi, D.N. / Pedersen, P.L. / Ko, Y.H. #1:  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 1998 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 1998タイトル: The 2.8-A structure of rat liver F1-ATPase: configuration of a critical intermediate in ATP synthesis/hydrolysis. 著者: Bianchet, M.A. / Hullihen, J. / Pedersen, P.L. / Amzel, L.M. #2: ジャーナル: Biochim.Biophys.Acta / 年: 1994 タイトル: The three-dimensional structure of rat liver mitochodria F1-ATPase: X-ray diffraction studies. 著者: Bianchet, M. / Medjahed, D. / Hulihen, J. / Pedersen, P.L. / Amzel, L.M. #3: ジャーナル: J.Bioenerg.Biomembr. / 年: 1992 タイトル: Quaternary structure of ATP synthases: symmetry and asymmetry in the F1 moiety. 著者: Amzel, L.M. / Bianchet, M.A. / Pedersen, P.L. #4: ジャーナル: J.Biol.Chem. / 年: 1991 タイトル: Mitochondrial ATP synthase. Quaternary structure of the F1 moiety at 3.6 A determined by x-ray diffraction analysis. 著者: Bianchet, M. / Ysern, X. / Hullihen, J. / Pedersen, P.L. / Amzel, L.M. | ||||||
| 履歴 |
| ||||||
| Remark 300 | BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 3 ... BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 3 CHAIN(S). SEE REMARK 350 FOR INFORMATION ON GENERATING THE BIOLOGICAL MOLECULE(S). THIS PROTEIN HAS AN UNUSUAL STOICHIOMETRY AND THUS, UNUSUAL SYMMETRY. THE BIOLOGICAL UNIT (ACTIVE ENZYME) IS COMPOSED OF 3 A CHAINS, 3 B CHAINS AND 1 G CHAIN. IN THE R32 SPACEGROUP OF THIS CRYSTAL, THE ASYMMETRIC UNIT IS MADE UP OF ONE A CHAIN, ONE B CHAIN AND ONE-THIRD OF A G CHAIN. THE G CHAIN LIES ON THE 3-FOLD CRYSTALLOGRAPHIC SYMMETRY AXIS AND EACH ASYMMETRIC UNIT CONTAINS A RANDOM ONE OF THE THREE CRYSTALLOGRAPHICALLY SYMMETRIC POSITIONS. TO MAKE A BIOMOLECULE, THE 3-FOLD MATRICES IN REMARK 350 ARE ONLY APPLIED TO CHAIN A AND CHAIN B. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2f43.cif.gz 2f43.cif.gz | 222.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2f43.ent.gz pdb2f43.ent.gz | 172.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2f43.json.gz 2f43.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2f43_validation.pdf.gz 2f43_validation.pdf.gz | 1008.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2f43_full_validation.pdf.gz 2f43_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  2f43_validation.xml.gz 2f43_validation.xml.gz | 53.4 KB | 表示 | |
| CIF形式データ |  2f43_validation.cif.gz 2f43_validation.cif.gz | 68.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/f4/2f43 https://data.pdbj.org/pub/pdb/validation_reports/f4/2f43 ftp://data.pdbj.org/pub/pdb/validation_reports/f4/2f43 ftp://data.pdbj.org/pub/pdb/validation_reports/f4/2f43 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1mabS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-ATP synthase ... , 3種, 3分子 ABG
| #1: タンパク質 | 分子量: 55334.195 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #2: タンパク質 | 分子量: 51404.461 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #3: タンパク質 | 分子量: 30231.725 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
-非ポリマー , 4種, 5分子 

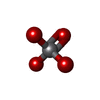




| #4: 化合物 | | #5: 化合物 | ChemComp-ATP / | #6: 化合物 | ChemComp-VO4 / | #7: 化合物 | ChemComp-ADP / | |
|---|
-詳細
| Has protein modification | N |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.1 Å3/Da / 溶媒含有率: 60 % |
|---|---|
| 結晶化 | 温度: 296 K / pH: 8.08 詳細: 5MM ATP, 5MM MGCL2, 5MM VANADATE, 25MM NAN3, 50MM MOPS, pH 8.08, VAPOR DIFFUSION, SITTING DROP, temperature 296K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.99092 / ビームライン: 19-ID / 波長: 0.99092 |
| 検出器 | タイプ: SBC-2 / 検出器: CCD / 詳細: MONOCROMATOR |
| 放射 | モノクロメーター: GRAPHITE / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.99092 Å / 相対比: 1 |
| 反射 | 解像度: 3→73.52 Å / Num. obs: 29647 / % possible obs: 100 % / 冗長度: 8.7 % / Rsym value: 0.143 / Net I/σ(I): 12.1 |
| 反射 シェル | 解像度: 3→3.16 Å / 冗長度: 8.9 % / Mean I/σ(I) obs: 3.2 / Rsym value: 0.75 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1MAB 解像度: 3→19.97 Å / Rfactor Rfree error: 0.008 / Data cutoff high absF: 7286337.69 / Data cutoff low absF: 0 / Isotropic thermal model: OVERALL / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 66.4837 Å2 / ksol: 0.277358 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 67.9 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3→19.97 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3→3.11 Å / Total num. of bins used: 10
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj






