| 登録情報 | データベース: PDB / ID: 2ci9
|
|---|
| タイトル | Nck1 SH2-domain in complex with a dodecaphosphopeptide from EPEC protein Tir |
|---|
 要素 要素 | - CYTOPLASMIC PROTEIN NCK1
- TRANSLOCATED INTIMIN RECEPTOR
|
|---|
 キーワード キーワード | PROTEIN BINDING / PROTEIN-BINDING / SH2-DOMAIN-COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of cap-dependent translational initiation / regulation of translation initiation in response to endoplasmic reticulum stress / eukaryotic initiation factor eIF2 binding / positive regulation of translation in response to endoplasmic reticulum stress / protein phosphatase type 1 complex / positive regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / positive regulation of cap-independent translational initiation / cytoskeletal anchor activity / substrate-dependent cell migration, cell extension / signal complex assembly ...positive regulation of cap-dependent translational initiation / regulation of translation initiation in response to endoplasmic reticulum stress / eukaryotic initiation factor eIF2 binding / positive regulation of translation in response to endoplasmic reticulum stress / protein phosphatase type 1 complex / positive regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / positive regulation of cap-independent translational initiation / cytoskeletal anchor activity / substrate-dependent cell migration, cell extension / signal complex assembly / Activation of RAC1 / Nephrin family interactions / DCC mediated attractive signaling / vesicle membrane / lamellipodium assembly / RHOV GTPase cycle / negative regulation of T cell receptor signaling pathway / protein kinase inhibitor activity / positive regulation of actin filament polymerization / negative regulation of PERK-mediated unfolded protein response / RHOU GTPase cycle / Generation of second messenger molecules / RHO GTPases Activate WASPs and WAVEs / ephrin receptor signaling pathway / ephrin receptor binding / signaling adaptor activity / positive regulation of T cell proliferation / antiviral innate immune response / Downstream signal transduction / SH2 domain binding / negative regulation of insulin receptor signaling pathway / protein sequestering activity / regulation of cell migration / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / response to endoplasmic reticulum stress / T cell activation / actin filament organization / FCGR3A-mediated phagocytosis / molecular condensate scaffold activity / positive regulation of neuron projection development / receptor tyrosine kinase binding / PKR-mediated signaling / Regulation of actin dynamics for phagocytic cup formation / VEGFA-VEGFR2 Pathway / cell-cell junction / cell migration / signaling receptor complex adaptor activity / protein-macromolecule adaptor activity / Potential therapeutics for SARS / ribosome / cadherin binding / signaling receptor binding / protein domain specific binding / lipid binding / host cell plasma membrane / endoplasmic reticulum / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / extracellular region / nucleus / membrane / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 Translocated intimin receptor, central domain / Translocated intimin receptor, N-terminal / Translocated intimin receptor / Translocated intimin receptor, C-terminal / Translocated intimin receptor, central domain superfamily / Translocated intimin receptor (Tir) intimin-binding domain / Translocated intimin receptor (Tir) C-terminus / Translocated intimin receptor (Tir) N-terminus / Nck1, SH3 domain 1 / Nck1, SH3 domain 2 ...Translocated intimin receptor, central domain / Translocated intimin receptor, N-terminal / Translocated intimin receptor / Translocated intimin receptor, C-terminal / Translocated intimin receptor, central domain superfamily / Translocated intimin receptor (Tir) intimin-binding domain / Translocated intimin receptor (Tir) C-terminus / Translocated intimin receptor (Tir) N-terminus / Nck1, SH3 domain 1 / Nck1, SH3 domain 2 / Nck1, SH3 domain 3 / Nck1, SH2 domain / Cytoplasmic protein NCK / : / SH2 domain / SHC Adaptor Protein / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / Src homology 3 domains / SH2 domain superfamily / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / SH3 domain / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Translocated intimin receptor Tir / Translocated intimin receptor Tir / SH2/SH3 adapter protein NCK1類似検索 - 構成要素 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト)
  ESCHERICHIA COLI (大腸菌) ESCHERICHIA COLI (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å |
|---|
 データ登録者 データ登録者 | Frese, S. / Schubert, W.-D. / Findeis, A.C. / Marquardt, T. / Roske, Y.S. / Stradal, T.E.B. / Heinz, D.W. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2006 ジャーナル: J.Biol.Chem. / 年: 2006
タイトル: The Phosphotyrosine Peptide Binding Specificity of Nck1 and Nck2 Src Homology 2 Domains.
著者: Frese, S. / Schubert, W.-D. / Findeis, A.C. / Marquardt, T. / Roske, Y.S. / Stradal, T.E.B. / Heinz, D.W. |
|---|
| 履歴 | | 登録 | 2006年3月17日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2006年4月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Refinement description / Version format compliance |
|---|
| 改定 1.2 | 2019年5月15日 | Group: Data collection / Derived calculations ...Data collection / Derived calculations / Experimental preparation / Other
カテゴリ: exptl_crystal_grow / pdbx_database_proc ...exptl_crystal_grow / pdbx_database_proc / pdbx_database_status / struct_conn
Item: _exptl_crystal_grow.method / _pdbx_database_status.recvd_author_approval / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 1.3 | 2024年5月1日 | Group: Data collection / Database references ...Data collection / Database references / Other / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf |
|---|
| 改定 1.4 | 2024年11月13日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. |
|---|
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR PROVIDED. |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト)
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.5 Å
分子置換 / 解像度: 1.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2006
ジャーナル: J.Biol.Chem. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2ci9.cif.gz
2ci9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2ci9.ent.gz
pdb2ci9.ent.gz PDB形式
PDB形式 2ci9.json.gz
2ci9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2ci9_validation.pdf.gz
2ci9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2ci9_full_validation.pdf.gz
2ci9_full_validation.pdf.gz 2ci9_validation.xml.gz
2ci9_validation.xml.gz 2ci9_validation.cif.gz
2ci9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ci/2ci9
https://data.pdbj.org/pub/pdb/validation_reports/ci/2ci9 ftp://data.pdbj.org/pub/pdb/validation_reports/ci/2ci9
ftp://data.pdbj.org/pub/pdb/validation_reports/ci/2ci9 リンク
リンク 集合体
集合体

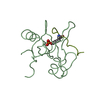
 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PGEX-6P-1 (AMERSHAM BIOSCIENCES) / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PGEX-6P-1 (AMERSHAM BIOSCIENCES) / 発現宿主: 

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト: MPG/DESY, HAMBURG
シンクロトロン / サイト: MPG/DESY, HAMBURG  / ビームライン: BW6 / 波長: 1.05
/ ビームライン: BW6 / 波長: 1.05  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














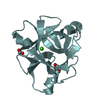
 PDBj
PDBj

















