+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2c7v | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of Trypanosoma brucei pteridine reductase (PTR1) in ternary complex with cofactor and the antifolate methotrexate | ||||||
 要素 要素 | PTERIDINE REDUCTASE | ||||||
 キーワード キーワード | OXIDOREDUCTASE / PTERIDINE REDUCTASE / TRYPANOSOMATIDS / DRUG RESISTANCE / SHORT-CHAIN DEHYDROGENASE/REDUCTASE / METHOTREXATE RESISTANCE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Dawson, A. / Gibellini, F. / Sienkiewicz, N. / Fyfe, P.K. / McLuskey, K. / Fairlamb, A.H. / Hunter, W.N. | ||||||
 引用 引用 |  ジャーナル: Mol.Microbiol. / 年: 2006 ジャーナル: Mol.Microbiol. / 年: 2006タイトル: Structure and Reactivity of Trypanosoma Brucei Pteridine Reductase: Inhibition by the Archetypal Antifolate Methotrexate 著者: Dawson, A. / Gibellini, F. / Sienkiewicz, N. / Tulloch, L.B. / Fyfe, P.K. / Mcluskey, K. / Fairlamb, A.H. / Hunter, W.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2c7v.cif.gz 2c7v.cif.gz | 228.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2c7v.ent.gz pdb2c7v.ent.gz | 184.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2c7v.json.gz 2c7v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2c7v_validation.pdf.gz 2c7v_validation.pdf.gz | 2.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2c7v_full_validation.pdf.gz 2c7v_full_validation.pdf.gz | 2.7 MB | 表示 | |
| XML形式データ |  2c7v_validation.xml.gz 2c7v_validation.xml.gz | 35.9 KB | 表示 | |
| CIF形式データ |  2c7v_validation.cif.gz 2c7v_validation.cif.gz | 51.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c7/2c7v https://data.pdbj.org/pub/pdb/validation_reports/c7/2c7v ftp://data.pdbj.org/pub/pdb/validation_reports/c7/2c7v ftp://data.pdbj.org/pub/pdb/validation_reports/c7/2c7v | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1e92S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||
| 単位格子 |
| ||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 28738.406 Da / 分子数: 4 / 由来タイプ: 組換発現 由来: (組換発現)  プラスミド: PET-TBPTR1H / 発現宿主:  |
|---|
-非ポリマー , 5種, 811分子 
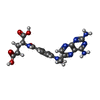







| #2: 化合物 | ChemComp-NAP / #3: 化合物 | ChemComp-MTX / #4: 化合物 | #5: 化合物 | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.01 Å3/Da / 溶媒含有率: 38.22 % |
|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 詳細: PROTEIN BUFFER WAS 20 MM TRIS HCL, PH 7.0 WITH 1MM NADP, 1 MM METHOTREXATE AND 20 MM DITHIOTHREITOL, PROTEIN CONCENTRATION WAS 6 MG/ML. HANGING DROP RESERVOIR SOLUTION WAS 0.1 M SODIUM ...詳細: PROTEIN BUFFER WAS 20 MM TRIS HCL, PH 7.0 WITH 1MM NADP, 1 MM METHOTREXATE AND 20 MM DITHIOTHREITOL, PROTEIN CONCENTRATION WAS 6 MG/ML. HANGING DROP RESERVOIR SOLUTION WAS 0.1 M SODIUM CACODYLATE PH 6.5 AND 1.4 M SODIUM ACETATE. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU IMAGE PLATE / 検出器: IMAGE PLATE / 日付: 2004年7月30日 / 詳細: MIRRORS |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→74.5 Å / Num. obs: 50048 / % possible obs: 99.2 % / 冗長度: 2.8 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 12.4 |
| 反射 シェル | 解像度: 2.2→2.26 Å / 冗長度: 2.1 % / Rmerge(I) obs: 0.17 / Mean I/σ(I) obs: 3.5 / % possible all: 93.1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PBD ENTRY 1E92 解像度: 2.2→74.54 Å / Cor.coef. Fo:Fc: 0.963 / Cor.coef. Fo:Fc free: 0.927 / SU B: 5.551 / SU ML: 0.142 / 交差検証法: THROUGHOUT / ESU R: 0.303 / ESU R Free: 0.218 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 24.22 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→74.54 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







