+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2bn2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
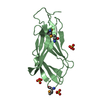
| タイトル | CRYSTAL STRUCTURE OF BOVINE NEUROPHYSIN II COMPLEXED WITH THE VASOPRESSIN ANALOGUE PHE-TYR AMIDE | |||||||||
 要素 要素 | NEUROPHYSIN II | |||||||||
 キーワード キーワード | HORMONE / HORMONE PACKAGING / TRANSPORT / PROTEIN-PEPTIDE COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報neurohypophyseal hormone activity / V1A vasopressin receptor binding / neuropeptide hormone activity / vasoconstriction / secretory granule / extracellular space 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / SAS / 解像度: 2.8 Å X線回折 / SAS / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Rose, J.P. / Wang, B.C. | |||||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1991 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1991タイトル: Crystal structure of a bovine neurophysin II dipeptide complex at 2.8 A determined from the single-wavelength anomalous scattering signal of an incorporated iodine atom. 著者: Chen, L.Q. / Rose, J.P. / Breslow, E. / Yang, D. / Chang, W.R. / Furey Jr., W.F. / Sax, M. / Wang, B.C. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1996 ジャーナル: Nat.Struct.Biol. / 年: 1996タイトル: Crystal Structure of the Neurophysin-Oxytocin Complex 著者: Rose, J.P. / Wu, C.K. / Hsiao, C.D. / Breslow, E. / Wang, B.C. #2:  ジャーナル: Eur.J.Biochem. / 年: 1988 ジャーナル: Eur.J.Biochem. / 年: 1988タイトル: Crystals of Modified Bovine Neurophysin II 著者: Rose, J.P. / Yang, D. / Yoo, C.S. / Sax, M. / Breslow, E. / Wang, B.C. #3:  ジャーナル: J.Mol.Biol. / 年: 1979 ジャーナル: J.Mol.Biol. / 年: 1979タイトル: Crystals of a Bovine Neurophysin II-Dipeptide Amide Complex 著者: Yoo, C.S. / Wang, B.C. / Sax, M. / Breslow, E. | |||||||||
| 履歴 |
| |||||||||
| Remark 650 | HELIX DETERMINATION METHOD: KSDSSP | |||||||||
| Remark 700 | SHEET DETERMINATION METHOD: KSDSSP |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2bn2.cif.gz 2bn2.cif.gz | 70.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2bn2.ent.gz pdb2bn2.ent.gz | 53.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2bn2.json.gz 2bn2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bn/2bn2 https://data.pdbj.org/pub/pdb/validation_reports/bn/2bn2 ftp://data.pdbj.org/pub/pdb/validation_reports/bn/2bn2 ftp://data.pdbj.org/pub/pdb/validation_reports/bn/2bn2 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 9890.252 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  #2: 化合物 | ChemComp-PHE / #3: 化合物 | ChemComp-TYR / Has protein modification | Y | 非ポリマーの詳細 | RESIDUES 98 AND 99 WITH CHAIN IDS A, C, E AND G FORM THE PHENYLALANINE-TYROSINE AMIDE DIPEPTIDE ...RESIDUES 98 AND 99 WITH CHAIN IDS A, C, E AND G FORM THE PHENYLALAN | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.82 Å3/Da / 溶媒含有率: 56 % 解説: SAS DATA WERE COLLECTED IN-HOUSE. THE ANOMALOUS SCATTERER WAS PARA IODO-PHENYLALANINE-TYROSINE AMIDE WHICH WAS BOUND IN THE HORMONE BINDING SITE. |
|---|---|
| 結晶化 | pH: 6.8 詳細: 5 MG OF PROTEIN WAS DISSOLVED IN 0.5 ML OF WATER, 0.5 MG OF PHENYLALANINE-TYROSINE AMIDE AND 20 MICRO LITERS OF SATURATED AMMONIUM SULPHATE SOLUTION WERE ADDED. THE PH OF THE SOLUTION WAS ADJUSTED TO 6.8 |
| 結晶化 | *PLUS pH: 7.5 / 手法: unknown |
| 溶液の組成 | *PLUS 一般名: ammonium sulfate |
-データ収集
| 回折 | 平均測定温度: 289 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 |
| 検出器 | タイプ: SIEMENS / 検出器: AREA DETECTOR / 日付: 1989年9月13日 / 詳細: SUPPER MIRRORS |
| 放射 | モノクロメーター: NI FILTER / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 最高解像度: 2.64 Å / Num. obs: 24898 / % possible obs: 83.6 % / Observed criterion σ(I): -3 / 冗長度: 17 % / Biso Wilson estimate: 60.2 Å2 / Rsym value: 0.0404 / Net I/σ(I): 17.05 |
| 反射 シェル | 解像度: 2.64→2.74 Å / Mean I/σ(I) obs: 3.12 / Rsym value: 0.1799 / % possible all: 43.45 |
| 反射 | *PLUS 最高解像度: 2.8 Å / Num. obs: 12840 / Num. measured all: 58983 / Rmerge(I) obs: 0.0577 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: SAS / 解像度: 2.8→8 Å / Rfactor Rfree error: 0.008 / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / Isotropic thermal model: OVERALL / 交差検証法: THROUGHOUT / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.7 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→8 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.97 Å / Rfactor Rfree error: 0.024 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.843 / 分類: refinement X-PLOR / バージョン: 3.843 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Num. reflection obs: 10729 / Rfactor obs: 0.225 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor obs: 0.298 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj