+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1xt7 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Daptomycin NMR Structure | ||||||
 要素 要素 | DAPTOMYCIN | ||||||
 キーワード キーワード | ANTIBIOTIC / DAPTOMYCIN / CUBICIN / LIPOPEPTIDE / CALCIUM-DEPENDENT | ||||||
| 機能・相同性 | Daptomycin / DECANOIC ACID / :  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  STREPTOMYCES ROSEOSPORUS (バクテリア) STREPTOMYCES ROSEOSPORUS (バクテリア) | ||||||
| 手法 | 溶液NMR / CONSTRAINED MOLECULAR DYNAMICS | ||||||
 データ登録者 データ登録者 | Ball, L.-J. / Goult, C.M. / Donarski, J.A. / Micklefield, J. / Ramesh, V. | ||||||
 引用 引用 |  ジャーナル: Org.Biomol.Chem. / 年: 2004 ジャーナル: Org.Biomol.Chem. / 年: 2004タイトル: NMR Structure Determination and Calcium Binding Effects of Lipopeptide Antibiotic Daptomycin 著者: Ball, L.-J. / Goult, C.M. / Donarski, J.A. / Micklefield, J. / Ramesh, V. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1xt7.cif.gz 1xt7.cif.gz | 13.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1xt7.ent.gz pdb1xt7.ent.gz | 7.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1xt7.json.gz 1xt7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1xt7_validation.pdf.gz 1xt7_validation.pdf.gz | 359.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1xt7_full_validation.pdf.gz 1xt7_full_validation.pdf.gz | 358.8 KB | 表示 | |
| XML形式データ |  1xt7_validation.xml.gz 1xt7_validation.xml.gz | 2.2 KB | 表示 | |
| CIF形式データ |  1xt7_validation.cif.gz 1xt7_validation.cif.gz | 2.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xt/1xt7 https://data.pdbj.org/pub/pdb/validation_reports/xt/1xt7 ftp://data.pdbj.org/pub/pdb/validation_reports/xt/1xt7 ftp://data.pdbj.org/pub/pdb/validation_reports/xt/1xt7 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 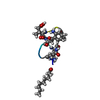
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド |   タイプ: Cyclic lipopeptide / クラス: 抗生剤 / 分子量: 1484.440 Da / 分子数: 1 / 由来タイプ: 合成 タイプ: Cyclic lipopeptide / クラス: 抗生剤 / 分子量: 1484.440 Da / 分子数: 1 / 由来タイプ: 合成詳細: DAPTOMYCIN IS AN ACIDIC CYCLIC LIPOPEPTIDE. THE SCAFFOLD IS MADE OF TWO PARTS: (1) THREE RESIDUES N-TERM EXOCYCLIC PART (2) A DECAPEPTIDE LACTONE RING DERIVED FROM CYCLISATION OF THR3 SIDE ...詳細: DAPTOMYCIN IS AN ACIDIC CYCLIC LIPOPEPTIDE. THE SCAFFOLD IS MADE OF TWO PARTS: (1) THREE RESIDUES N-TERM EXOCYCLIC PART (2) A DECAPEPTIDE LACTONE RING DERIVED FROM CYCLISATION OF THR3 SIDE CHAIN ONTO THE C-TER CARBOXYL GROUP THE N-DECANOYL FATTY ACID IS LINKED TO THE MAIN BODY OF THE MOLECULE VIA N-TERM ACYLATION. 由来: (合成)  STREPTOMYCES ROSEOSPORUS (バクテリア) / 参照: NOR: NOR00001, Daptomycin STREPTOMYCES ROSEOSPORUS (バクテリア) / 参照: NOR: NOR00001, Daptomycin |
|---|---|
| #2: 化合物 | ChemComp-DKA / 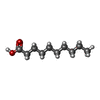  タイプ: Cyclic lipopeptide / クラス: 抗生剤 / 分子量: 172.265 Da / 分子数: 1 / 由来タイプ: 合成 / 式: C10H20O2 タイプ: Cyclic lipopeptide / クラス: 抗生剤 / 分子量: 172.265 Da / 分子数: 1 / 由来タイプ: 合成 / 式: C10H20O2詳細: DAPTOMYCIN IS AN ACIDIC CYCLIC LIPOPEPTIDE. THE SCAFFOLD IS MADE OF TWO PARTS: (1) THREE RESIDUES N-TERM EXOCYCLIC PART (2) A DECAPEPTIDE LACTONE RING DERIVED FROM CYCLISATION OF THR3 SIDE ...詳細: DAPTOMYCIN IS AN ACIDIC CYCLIC LIPOPEPTIDE. THE SCAFFOLD IS MADE OF TWO PARTS: (1) THREE RESIDUES N-TERM EXOCYCLIC PART (2) A DECAPEPTIDE LACTONE RING DERIVED FROM CYCLISATION OF THR3 SIDE CHAIN ONTO THE C-TER CARBOXYL GROUP THE N-DECANOYL FATTY ACID IS LINKED TO THE MAIN BODY OF THE MOLECULE VIA N-TERM ACYLATION. 参照: Daptomycin |
| 構成要素の詳細 | DAPTOMYCIN IS A CYCLIC TRIDECAMER LIPOPETIDE. HERE, DAPTOMYCIN IS REPRESENTED BY GROUPING TOGETHER ...DAPTOMYCIN |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | タイプ: 2D NOESY |
| NMR実験の詳細 | Text: THIS STRUCTURE WAS DETERMINED USING STANDARD 2D HOMONUCLEAR TECHNIQUES. |
- 試料調製
試料調製
| 詳細 |
| ||||||
|---|---|---|---|---|---|---|---|
| 試料状態 | pH: 5.05 / 圧: AMBIENT / 温度: 298 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Bruker DRX / 製造業者: Bruker / モデル: DRX / 磁場強度: 600 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: CONSTRAINED MOLECULAR DYNAMICS / ソフトェア番号: 1 詳細: A TOTAL OF 52 DISTANCE RESTRAINTS WERE USED FOR NMR STRUCTURE CALCULATION. | ||||||||||||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj