| 登録情報 | データベース: PDB / ID: 1xpa
|
|---|
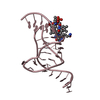
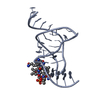
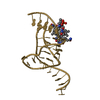
| タイトル | SOLUTION STRUCTURE OF THE DNA-AND RPA-BINDING DOMAIN OF THE HUMAN REPAIR FACTOR XPA, NMR, 1 STRUCTURE |
|---|
 要素 要素 | XPA |
|---|
 キーワード キーワード | DNA REPAIR / NUCLEOTIDE EXCISION REPAIR / ZINC-FINGER |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
nucleotide-excision repair factor 1 complex / nucleotide-excision repair involved in interstrand cross-link repair / nucleotide-excision repair, DNA damage recognition / response to auditory stimulus / UV protection / UV-damage excision repair / intercellular bridge / protein localization to nucleus / nucleotide-excision repair / base-excision repair ...nucleotide-excision repair factor 1 complex / nucleotide-excision repair involved in interstrand cross-link repair / nucleotide-excision repair, DNA damage recognition / response to auditory stimulus / UV protection / UV-damage excision repair / intercellular bridge / protein localization to nucleus / nucleotide-excision repair / base-excision repair / Formation of TC-NER Pre-Incision Complex / Dual Incision in GG-NER / Formation of Incision Complex in GG-NER / Dual incision in TC-NER / sequence-specific double-stranded DNA binding / damaged DNA binding / protein domain specific binding / DNA repair / protein homodimerization activity / nucleoplasm / nucleus / metal ion binding類似検索 - 分子機能 Nucleotide Excision Repair Protein XPA (XPA-MBD); B Chain A / XPA C-terminal domain / XPA protein N-terminal / XPA / Zinc finger, XPA-type, conserved site / XPA, C-terminal / XPA, conserved site / XPA protein C-terminus / XPA protein signature 1. / XPA protein signature 2. ...Nucleotide Excision Repair Protein XPA (XPA-MBD); B Chain A / XPA C-terminal domain / XPA protein N-terminal / XPA / Zinc finger, XPA-type, conserved site / XPA, C-terminal / XPA, conserved site / XPA protein C-terminus / XPA protein signature 1. / XPA protein signature 2. / XPA domain superfamily / Putative DNA-binding domain superfamily / Alpha-Beta Complex / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / distance geometry |
|---|
 データ登録者 データ登録者 | Ikegami, T. / Kuraoka, I. / Saijo, M. / Kodo, N. / Kyogoku, Y. / Morikawa, K. / Tanaka, K. / Shirakawa, M. |
|---|
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1998 ジャーナル: Nat.Struct.Biol. / 年: 1998
タイトル: Solution structure of the DNA- and RPA-binding domain of the human repair factor XPA.
著者: Ikegami, T. / Kuraoka, I. / Saijo, M. / Kodo, N. / Kyogoku, Y. / Morikawa, K. / Tanaka, K. / Shirakawa, M. |
|---|
| 履歴 | | 登録 | 1998年7月6日 | 処理サイト: BNL |
|---|
| 改定 1.0 | 1999年7月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年3月24日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年3月2日 | Group: Database references / Derived calculations / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_struct_assembly / pdbx_struct_conn_angle / pdbx_struct_oper_list / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.process_site / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Biol. / 年: 1998
ジャーナル: Nat.Struct.Biol. / 年: 1998 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1xpa.cif.gz
1xpa.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1xpa.ent.gz
pdb1xpa.ent.gz PDB形式
PDB形式 1xpa.json.gz
1xpa.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1xpa_validation.pdf.gz
1xpa_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1xpa_full_validation.pdf.gz
1xpa_full_validation.pdf.gz 1xpa_validation.xml.gz
1xpa_validation.xml.gz 1xpa_validation.cif.gz
1xpa_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xp/1xpa
https://data.pdbj.org/pub/pdb/validation_reports/xp/1xpa ftp://data.pdbj.org/pub/pdb/validation_reports/xp/1xpa
ftp://data.pdbj.org/pub/pdb/validation_reports/xp/1xpa リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / プラスミド: PET16B / 生物種 (発現宿主): Escherichia coli / 遺伝子 (発現宿主): HUMAN XPA CDNA / 発現宿主:
Homo sapiens (ヒト) / プラスミド: PET16B / 生物種 (発現宿主): Escherichia coli / 遺伝子 (発現宿主): HUMAN XPA CDNA / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj



 X-PLOR
X-PLOR