+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1xft | ||||||
|---|---|---|---|---|---|---|---|
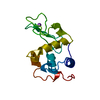
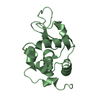
| タイトル | Synchrotron X-ray Powder Diffraction Study of Hexagonal Turkey Egg-white Lysozyme | ||||||
 要素 要素 | Lysozyme C | ||||||
 キーワード キーワード | HYDROLASE / powder diffraction / lysozyme / x-rays | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glycosaminoglycan binding / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / defense response to Gram-negative bacterium / killing of cells of another organism / defense response to Gram-positive bacterium / extracellular space / identical protein binding / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 粉末回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.35 Å 分子置換 / 解像度: 3.35 Å | ||||||
 データ登録者 データ登録者 | Margiolaki, I. / Wright, J.P. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005タイトル: Synchrotron X-ray powder diffraction study of hexagonal turkey egg-white lysozyme. 著者: Margiolaki, I. / Wright, J.P. / Fitch, A.N. / Fox, G.C. / Von Dreele, R.B. #1:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2001 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2001タイトル: Binding of N-acetylglucosamine to chicken egg lysozyme: a powder diffraction study 著者: Von Dreele, R.B. #2:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1995 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1995タイトル: Structure of hexagonal turkey egg-white lysozyme at 1.65A resolution 著者: Howell, P.L. #3:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1993 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1993タイトル: X-ray structure of monoclinic turkey egg lysozyme at 1.3 A resolution 著者: Harata, K. #4:  ジャーナル: Acta Crystallogr.,Sect.B / 年: 1992 ジャーナル: Acta Crystallogr.,Sect.B / 年: 1992タイトル: Structure determination of turkey egg-white lysozyme using Laue diffraction data 著者: Howell, P.L. / Almo, S.C. / Parsons, M.R. / Hajdu, J. / Petsko, G.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1xft.cif.gz 1xft.cif.gz | 33.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1xft.ent.gz pdb1xft.ent.gz | 19.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1xft.json.gz 1xft.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xf/1xft https://data.pdbj.org/pub/pdb/validation_reports/xf/1xft ftp://data.pdbj.org/pub/pdb/validation_reports/xf/1xft ftp://data.pdbj.org/pub/pdb/validation_reports/xf/1xft | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1tewS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14228.105 Da / 分子数: 1 / 断片: LYSOZYME / 由来タイプ: 天然 / 詳細: sigma-aldrich chemical company LOT. 064H7230 由来: (天然)  組織: egg white / 参照: UniProt: P00703, lysozyme |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 粉末回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.25 Å3/Da / 溶媒含有率: 44.81 % |
|---|---|
| 結晶化 | 温度: 295 K / 手法: salting out / pH: 6 / 詳細: NaCl, pH 6, SALTING OUT, temperature 295K |
-データ収集
| 回折 | 平均測定温度: 295 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID31 / 波長: 0.700667 Å / ビームライン: ID31 / 波長: 0.700667 Å |
| 検出器 | タイプ: CUSTOM-MADE / 検出器: DIFFRACTOMETER / 日付: 2003年12月3日 詳細: DOUBLE SI(111) MONOCHROMATOR, 9 CHANNEL SI(111) MULTI-ANALYSER |
| 放射 | モノクロメーター: DOUBLE SI(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.700667 Å / 相対比: 1 |
| 反射 | 解像度: 3.35→95.1 Å / Num. all: 2099 / Num. obs: 2099 / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1TEW 解像度: 3.35→95.1 Å / Isotropic thermal model: OVERALL / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber / 詳細: POWDER DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.69 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.35→95.1 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj
