登録情報 データベース : PDB / ID : 1u0fタイトル Crystal structure of mouse phosphoglucose isomerase in complex with glucose 6-phosphate Glucose-6-phosphate isomerase キーワード / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / / 解像度 : 1.6 Å データ登録者 Solomons, J.T.G. / Zimmerly, E.M. / Burns, S. / Krishnamurthy, N. / Swan, M.K. / Krings, S. / Muirhead, H. / Chirgwin, J. / Davies, C. ジャーナル : J.Mol.Biol. / 年 : 2004タイトル : The crystal structure of mouse phosphoglucose isomerase at 1.6A resolution and its complex with glucose 6-phosphate reveals the catalytic mechanism of sugar ring opening.著者 : Graham Solomons, J.T. / Zimmerly, E.M. / Burns, S. / Krishnamurthy, N. / Swan, M.K. / Krings, S. / Muirhead, H. / Chirgwin, J. / Davies, C. 履歴 登録 2004年7月13日 登録サイト / 処理サイト 改定 1.0 2004年11月2日 Provider / タイプ 改定 1.1 2008年4月30日 Group 改定 1.2 2011年7月13日 Group / Version format compliance改定 1.3 2017年10月11日 Group / カテゴリ 改定 1.4 2020年7月29日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Structure summary カテゴリ chem_comp / entity ... chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_ref_seq_dif / struct_site / struct_site_gen Item _chem_comp.mon_nstd_flag / _chem_comp.name ... _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_ref_seq_dif.details 解説 / Provider / タイプ 改定 1.5 2024年2月14日 Group / Database references / Structure summaryカテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession改定 1.6 2024年4月3日 Group / カテゴリ
すべて表示 表示を減らす Remark 999 SEQUENCE RESIDUE 263 IS A LEU NOT A PHE AS SHOWN BY THE SEQUENCE OF THE CONSTRUCT AS WELL AS THE ... SEQUENCE RESIDUE 263 IS A LEU NOT A PHE AS SHOWN BY THE SEQUENCE OF THE CONSTRUCT AS WELL AS THE ELECTRON DENSITY MAP. IT APPEARS TO EMANATE FROM THE ORIGINAL EXPRESSED SEQUENCE TAG AND IS NOT A PCR ERROR. HENCE THIS IS A POLYMORPHISM IN THE MAMMARY CELL LINE USED TO MAKE THE CDNA.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン / REFINEMENT FROM THE NATIVE STRUCTURE / 解像度: 1.6 Å
シンクロトロン / REFINEMENT FROM THE NATIVE STRUCTURE / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2004
ジャーナル: J.Mol.Biol. / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1u0f.cif.gz
1u0f.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1u0f.ent.gz
pdb1u0f.ent.gz PDB形式
PDB形式 1u0f.json.gz
1u0f.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/u0/1u0f
https://data.pdbj.org/pub/pdb/validation_reports/u0/1u0f ftp://data.pdbj.org/pub/pdb/validation_reports/u0/1u0f
ftp://data.pdbj.org/pub/pdb/validation_reports/u0/1u0f リンク
リンク 集合体
集合体
 要素
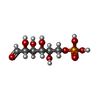
要素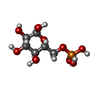











 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-ID / 波長: 1 Å
/ ビームライン: 22-ID / 波長: 1 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj

















