+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1qcu | ||||||
|---|---|---|---|---|---|---|---|
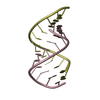
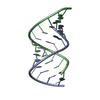
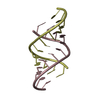
| タイトル | CRYSTAL STRUCTURE OF AN 18 BASE PAIR COPY CONTROL RELATED RNA DUPLEX | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA / A-RNA STRUCTURE / RIBONUCLEIC ACID | ||||||
| 機能・相同性 | AMMONIUM ION / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.2 Å 分子置換 / 解像度: 1.2 Å | ||||||
 データ登録者 データ登録者 | Klosterman, P.S. / Shah, S.A. / Steitz, T.A. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1999 ジャーナル: Biochemistry / 年: 1999タイトル: Crystal structures of two plasmid copy control related RNA duplexes: An 18 base pair duplex at 1.20 A resolution and a 19 base pair duplex at 1.55 A resolution. 著者: Klosterman, P.S. / Shah, S.A. / Steitz, T.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1qcu.cif.gz 1qcu.cif.gz | 74.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1qcu.ent.gz pdb1qcu.ent.gz | 59 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1qcu.json.gz 1qcu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1qcu_validation.pdf.gz 1qcu_validation.pdf.gz | 417.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1qcu_full_validation.pdf.gz 1qcu_full_validation.pdf.gz | 426.9 KB | 表示 | |
| XML形式データ |  1qcu_validation.xml.gz 1qcu_validation.xml.gz | 8.1 KB | 表示 | |
| CIF形式データ |  1qcu_validation.cif.gz 1qcu_validation.cif.gz | 10.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qc/1qcu https://data.pdbj.org/pub/pdb/validation_reports/qc/1qcu ftp://data.pdbj.org/pub/pdb/validation_reports/qc/1qcu ftp://data.pdbj.org/pub/pdb/validation_reports/qc/1qcu | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 3752.306 Da / 分子数: 2 / 由来タイプ: 合成 #2: RNA鎖 | 分子量: 3312.042 Da / 分子数: 2 / 由来タイプ: 合成 #3: 化合物 | ChemComp-NH4 / #4: 水 | ChemComp-HOH / | 構成要素の詳細 | THE CRYSTALS EXHIBIT 36-FOLD STATIC DISORDER, SUCH THAT THERE IS ONE CONFORMATION OF THE SUGAR- ...THE CRYSTALS EXHIBIT 36-FOLD STATIC DISORDER, SUCH THAT THERE IS ONE CONFORMATI | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 2 X線回折 / 使用した結晶の数: 2 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 49.5 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 詳細: (NH4)2SO4, SODIUM CACODYLATE, MGCL2, ROP PROTEIN, pH 6.5, VAPOR DIFFUSION, HANGING DROP, temperature 293K | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ / 手法: 蒸気拡散法 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| ||||||||||||||||||
| 検出器 |
| ||||||||||||||||||
| 放射 |
| ||||||||||||||||||
| 放射波長 |
| ||||||||||||||||||
| 反射 | 解像度: 1.2→30 Å / Num. all: 19666 / Num. obs: 18266 / % possible obs: 92.5 % / Observed criterion σ(F): -3 / Observed criterion σ(I): -3 / 冗長度: 4.2 % / Biso Wilson estimate: 21.7 Å2 / Rmerge(I) obs: 0.073 / Net I/σ(I): 31.8 | ||||||||||||||||||
| 反射 シェル | 解像度: 1.2→1.24 Å / 冗長度: 2.8 % / Rmerge(I) obs: 0.274 / Mean I/σ(I) obs: 5.2 / % possible all: 77.1 | ||||||||||||||||||
| 反射 | *PLUS % possible obs: 93 % | ||||||||||||||||||
| 反射 シェル | *PLUS % possible obs: 77 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: A-RNA 解像度: 1.2→30 Å / Num. parameters: 7977 / Num. restraintsaints: 21649 / 交差検証法: FREE R / σ(F): 0 / σ(I): 0 StereochEM target val spec case: USED THE AVERAGE OF GUA C6 O6, ADE C6 N6 BOND LENGTHS AS THE C6 O6 BOND LENGTH 立体化学のターゲット値: Parkinson et al. 詳細: ANISOTROPIC REFINEMENT REDUCED FREE R (NO CUTOFF) BY 6.5% THE MOLECULE CRYSTALLIZED IN SPACE GROUP P321. HOWEVER THE REFINEMENT WAS CARRIED OUT IN THE LOWER SPACE GROUP P3. THE COORDINATES ...詳細: ANISOTROPIC REFINEMENT REDUCED FREE R (NO CUTOFF) BY 6.5% THE MOLECULE CRYSTALLIZED IN SPACE GROUP P321. HOWEVER THE REFINEMENT WAS CARRIED OUT IN THE LOWER SPACE GROUP P3. THE COORDINATES DEPOSITED ARE FOR THE SPACE GROUP P3.
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL., 91 201-228 (1973) | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 22 / Occupancy sum hydrogen: 227 / Occupancy sum non hydrogen: 578 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.2→30 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor all: 0.202 / Rfactor obs: 0.116 / Rfactor Rfree: 0.15 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj


































