+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1pfe | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Echinomycin-(gcgtacgc)2 complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA/ANTIBIOTIC / BISINTERCALATOR / HOOGSTEEN BASEPAIR / DEPSIPEPTIDE / QUINOXALINE / THIOACETAL / ANTIBIOTIC / ANTITUMOR / DNA-ANTIBIOTIC COMPLEX | ||||||
| 機能・相同性 | Echinomycin / 2-CARBOXYQUINOXALINE / : / DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  STREPTOMYCES ECHINATUS (バクテリア) STREPTOMYCES ECHINATUS (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.1 Å 分子置換 / 解像度: 1.1 Å | ||||||
 データ登録者 データ登録者 | Cuesta-Seijo, J.A. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005タイトル: Structures of Complexes between Echinomycin and Duplex DNA. 著者: Cuesta-Seijo, J.A. / Sheldrick, G.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1pfe.cif.gz 1pfe.cif.gz | 30.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1pfe.ent.gz pdb1pfe.ent.gz | 20.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1pfe.json.gz 1pfe.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1pfe_validation.pdf.gz 1pfe_validation.pdf.gz | 414.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1pfe_full_validation.pdf.gz 1pfe_full_validation.pdf.gz | 418 KB | 表示 | |
| XML形式データ |  1pfe_validation.xml.gz 1pfe_validation.xml.gz | 5.1 KB | 表示 | |
| CIF形式データ |  1pfe_validation.cif.gz 1pfe_validation.cif.gz | 6.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pf/1pfe https://data.pdbj.org/pub/pdb/validation_reports/pf/1pfe ftp://data.pdbj.org/pub/pdb/validation_reports/pf/1pfe ftp://data.pdbj.org/pub/pdb/validation_reports/pf/1pfe | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||
| 単位格子 |
| |||||||||||||||
| Components on special symmetry positions |
| |||||||||||||||
| 詳細 | The second part of the biological assembly is generated by: -x, -x+y, -z |
- 要素
要素
| #1: DNA鎖 | 分子量: 2427.605 Da / 分子数: 1 / 由来タイプ: 合成 | ||||
|---|---|---|---|---|---|
| #2: タンパク質・ペプチド |   タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 809.008 Da / 分子数: 1 / 由来タイプ: 天然 タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 809.008 Da / 分子数: 1 / 由来タイプ: 天然詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 由来: (天然)  STREPTOMYCES ECHINATUS (バクテリア) / 参照: NOR: NOR01126, Echinomycin STREPTOMYCES ECHINATUS (バクテリア) / 参照: NOR: NOR01126, Echinomycin | ||||
| #3: 化合物 | ChemComp-CL / | ||||
| #4: 化合物 | 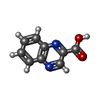  タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2 タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 参照: Echinomycin #5: 水 | ChemComp-HOH / | 構成要素の詳細 | THE ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE, A MEMBER OF THE QUINOXALINE CLASS OF ANTIBIOTICS. ...THE ECHINOMYCI | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.51 Å3/Da / 溶媒含有率: 64.98 % | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 4.5 詳細: LI2SO4, MGCL2, METHANOL, NAAC, SPERMINE, PH 4.5, VAPOR DIFFUSION, HANGING DROP, TEMPERATURE 293K | ||||||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: X11 / 波長: 0.8162 / ビームライン: X11 / 波長: 0.8162 |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2002年11月19日 |
| 放射 | モノクロメーター: TRIANGULAR MONOCHROMATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.8162 Å / 相対比: 1 |
| 反射 | 解像度: 1.1→21 Å / Num. obs: 15164 / % possible obs: 97.2 % / Observed criterion σ(I): 0 / 冗長度: 14.91 % / Rmerge(I) obs: 0.049 / Net I/σ(I): 30.01 |
| 反射 シェル | 解像度: 1.1→1.2 Å / 冗長度: 13.05 % / Rmerge(I) obs: 0.242 / Mean I/σ(I) obs: 11.48 / % possible all: 95.7 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: TROSTIN A - (GCGTACGC)2 COMPLEX 解像度: 1.1→21 Å / Num. parameters: 2979 / Num. restraintsaints: 2074 / 交差検証法: FREE R / σ(F): 0 StereochEM target val spec case: TARGETS FOR ECHINOMYCIN BUILT FROM THE CSD 立体化学のターゲット値: STANDARD SHELX FOR THE DNA, ENGH & HUBER FOR THE STANDARD PROTEIN PARTS OF ECHINOMYCIN, BUILT FROM THE CSD FOR THE REST 詳細: THE SKELETON OF ECHINOMYCIN IS NEARLY SYMMETRIC (NOT MODELLED TO DO SO), THE BRIDGE IS NOT. BOTH POSSIBLE ORIENTATIONS OF BINDING WERE OBSERVED, BUT SKELETON POSITIONS NEARLY OVERLAP, SO ONLY ...詳細: THE SKELETON OF ECHINOMYCIN IS NEARLY SYMMETRIC (NOT MODELLED TO DO SO), THE BRIDGE IS NOT. BOTH POSSIBLE ORIENTATIONS OF BINDING WERE OBSERVED, BUT SKELETON POSITIONS NEARLY OVERLAP, SO ONLY THE BRIDGE WAS MODELLED AS DISORDERED/MICROHETEROGENEITY.
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 7 / Occupancy sum hydrogen: 149 / Occupancy sum non hydrogen: 303 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.1→21 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.1→1.2 Å / % reflection obs: 95.7 % |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj


